Uno de los avances clave de la enología moderna es el reconocimiento de la importancia de la levadura como un agente imprescindible para la adecuada obtención de vino y cava. De este reconocimiento nace, de manera inmediata, la necesidad de controlar las propiedades genéticas y metabólicas de las cepas empleadas para las fermentaciones enológicas, así como la de desarrollar sistemas analíticos capaces de distinguir entre las diferentes cepas de levadura, tanto por las propias (o deseadas) como las contaminantes.
Actualmente existen varios métodos de identificación de levaduras de interés enológico para técnicas de biología molecular con resultados rápidos, fiables y económicos. Estos métodos se basan esencialmente en el análisis directo del DNA de la levadura; las diferencias entre las propiedades de este DNA se denominan polimorfismo.
El mayor reto al identificar cepas de levadura recae en la necesidad de diferenciar grupos y especies que taxonómicamente están muy cerca, pero que tienen propiedades muy diferentes en lo que respecta a sus aptitudes fermentativas y organolépticas. Además, durante las primeras fases de la fermentación aparecen en el mosto levaduras de los géneros Kloeckera, Candida, Rhodotorula, Pichia, Kluyveromyces o Hansenula, entre otros. La presencia de estas especies puede dar lugar a metabolitos secundarios que modifiquen las propiedades organolépticas del producto final, y, por tanto, es importante disponer de buenos métodos de detección para su control. Finalmente, también es posible utilizar las técnicas de identificación molecular para el análisis y comprobación de la estabilidad genética de las cepas de levadura, un aspecto importante cuando se manejan grandes volúmenes de cultivo, como es el caso de los tirajes de las grandes firmas del cava.
Determinación de especie
Taxonómicamente, las levaduras que se añaden a la harina para que «suba» (de ahí el término levadura, del latín levare, levantar), al maltear para que dé cerveza (de aquí cerevisiae, de la cerveza) y las que convierten el mosto en vino, pertenecen a una misma especie, Saccharomyces cerevisiae. Asimismo, las levaduras que añadimos juntamente con el azúcar al vino para dar cava y las que forman el velo que origina el jerez y vinos similares son de la misma especie. Lo que ahora conocemos como género Saccharomyces ha sido subdividido y reagrupado en diversas ocasiones desde que se definió en 1870. Hoy en día, se considera que este género tiene 10 especies, las más importante de las cuales es precisamente S. cerevisiae. A pesar de ello, dentro de esta especie existe una gran diversidad, que obliga a subdividirla en seis grupos, sin valor taxonómico, aunque con relevancia práctica. Estos grupos (llamados cerevisiae, ellipsoideus, oviformis, cheresanus, diastaticus y logos) más o menos coinciden con los diferentes usos de estas variedades. Por ahora, el sistema más fiable de clasificar las levaduras se basa en comparaciones directas de las composiciones de los DNA de la cepa problema con una cepa patrón, por hibridación. Se considera que dos variedades pertenecen a la misma especie cuando sus DNA difieren en menos del 20%. Como comparación podemos pensar que diferentes secuencias de DNA entre Homo sapiens (en humanos) y Pan troglodytes (chimpancé) son sólo del 3 al 5%. De todas maneras, este método es muy complicado y sólo está al alcance de laboratorios muy especializados.
Otro sistema para diferenciar entre géneros y especies distintas dentro de las levaduras del vino es el análisis de los polimorfismos en el DNA ribosomal. Como todos los eucariotas, las levaduras tienen un subcompartimiento del núcleo, dedicado a la síntesis y ensamblaje de los ribosomas, que son la maquinaria transcripcional de la célula. La producción de ribosomas, que contienen un total de 77 proteínas diferentes además de los cuatro RNA ribosomales (rRNA), ocurre en la levadura a velocidades realmente vertiginosas: se calcula que una cepa de Saccharomyces cerevisiae de laboratorio fabrica un máximo de cuatro ribosomas por segundo. Parte de esta velocidad se debe a la disposición de los genes que codifican por los RNA ribosomales al genoma: los cuatro genes se encuentran contenidos en el genoma, creando un paquete (un repeat) de unas 9 kb (millares de pares de bases), y esta subunidad básica se encuentra repetida entre 50 y 200 veces. Mientras que las secuencias que codifican por los diferentes rRNA se encuentran extraordinariamente conservadas entre especies e incluso entre géneros, familias y órdenes diferentes, las zonas de DNA entre estas secuencias codificantes, denominadas ITS (de Internal Transcribed Spacers, secuencias internas transcritas) son zonas muy poco conservadas. Precisamente, las ITS son utilizadas para diferenciar entre especies de Saccharomyces y entre diferentes géneros de levaduras. Metodológicamente, lo que se hace es amplificar DNA de estas regiones por la técnica de la PCR (reacción en cadena de la polimerasa de DNA) a partir de DNA parcialmente purificado y oligonucleótidos cebadores especialmente diseñados, como se muestra en la figura 1.
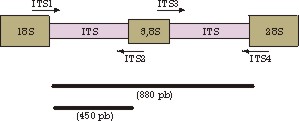
Tanto por las medidas de los amplificados como por los fragmentos que resultan de su digestión con diferentes enzimas de restricción, RFLP de rRNA (del inglés, Restriction Fragment Length Polymorphism, RFLP, polimorfismo en la longitud de los fragmentos de restricción), se puede distinguir entre levaduras de diferentes géneros o de diferentes especies del género Saccharomyces. Potencialmente, se trata de un sistema muy sensible de detección de contaminantes en las fermentaciones. Aunque este método pueda parecer complicado conceptualmente, el utillaje y las técnicas de PCR están tan normalizadas que lo ponen al alcance de un laboratorio medianamente equipado.
Identificación de subespecies y cepas
El genoma de la levadura, recientemente secuenciado en su totalidad, tiene un total de 14 Mb (millones de pares de bases) en su dotación haploide, repartidas entre 16 cromosomas, de una medida que oscila entre 150 y 2000 kb. La visualización de los cromosomas de una especie, que a menudo se hace por electroforesis de campo pulsante, recibe el nombre de cariotipo (figura 2).
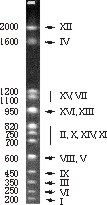
El cariotipo permite distinguir entre diferentes especies del género Saccharomyces y entre éste y otros géneros. Además, y según nuestros datos, los cariotipos de poblaciones naturales de S. cerevisiae del mosto son sorprendentemente elevados, lo que sugiere que sólo cepas muy próximas presentan el mismo cariotipo. Hay que decir que no es un fenómeno universal: las cepas de levadura flor, utilizadas para elaborar el vino de jerez, presentan cariotipos muy constantes. Si nos limitamos a la levadura de la fermentación del mosto, este método permite reconocer el grado de parentesco entre cepas procedentes de diferentes instalaciones, independientemente de su morfología o de su comportamiento metabólico. De todas formas, el análisis cariotípico por campo pulsante es caro, complicado y lento, difícil de adaptarse a las necesidades cotidianas de una bodega. Sea como sea, se trata de una herramienta valiosísima, a la que volveremos a referirnos mas adelante, cuando tratemos el tema de la inestabilidad genética.
La técnica posiblemente más sencilla y eficaz para distinguir diferentes cepas de levadura no se basa en el análisis del DNA cromosómico, sino del DNA mitocondrial (mtDNA). La mitocondria, la fábrica energética de los eucariotas, contiene como un relicto de su origen procariótico una molécula de DNA circular, que en la levadura tiene unos 75 kb. Esta molécula presenta una gran variabilidad de secuencia, lo que la convierte en un instrumento valioso para la identificación de cepas de levadura. Además, y debido a la diferente composición en pares de bases entre el mtDNA y el DNA genómico, es posible hacer digestiones enzimáticas que corten completamente este último, a pesar de producir bandas de medidas discretas provinentes exclusivamente del mtDNA (RFLP de mtDNA). Las medidas de estos fragmentos de restricción varían entre los diferentes géneros de levaduras, entre las diferentes especies y grupos de Saccharomyces, y entre diferentes clones de S. cerevisiae.
Por ejemplo, en la figura 3 se muestran 15 patrones de corte con el enzima HinfI procedentes del mtDNA de cepas aisladas del mosto durante dos años consecutivos (1994 y1995) en una misma bodega del Penedès. La gran variabilidad hace que este análisis permita comprobar la pureza de un cultivo o la implantación de una cepa seleccionada en una fermentación enológica, ambas de gran importancia en los procesos industriales de elaboración tanto de vino como de cava.
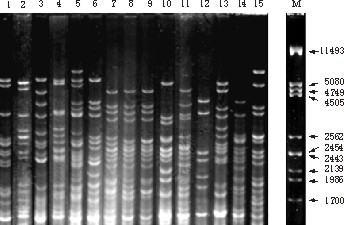
Variabilidad genética o qué es un clon
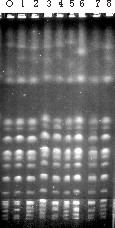
Así como resulta difícil definir el concepto de especie en los microorganismos, tampoco el concepto de clon es fácilmente determinable. Un clon es un conjunto de individuos (de células, en este caso) que proviene de una única célula original. El concepto de clonación es uno de los más importantes en microbiología (y no tan sólo en microbiología), ya que asegura que todos los individuos (células) clónicos son idénticos entre sí. Este concepto, que funciona perfectamente en laboratorio, pierde parte de su sentido cuando nos enfrentamos a cepas de levaduras naturales o industriales. Por mecanismos que todavía se desconocen, células de levaduras provenientes de un único origen clonal pueden desarrollar características nuevas simplemente por crecimiento vegetativo, sin reproducción sexual. Aunque este fenómeno hoy por hoy no es controlable, lo que sí se puede hacer es observar algunos de sus efectos: el más aparente es el conjunto de cambios en el cariotipo.
En la figura 4 se muestra un ejemplo de variabilidad en el cariotipo al cabo de 100 doblajes de un único clon (el marcado como O en la figura) en un medio de laboratorio. Los ocho clones que se presentan no han sido seleccionados, sino que correspondes a una muestra al azar. Se puede comprobar que ninguno de los clones aislados después de 100 generaciones mantiene el cariotipo original. Con más o menos frecuencia, este fenómeno pasa en la mayoría, sino en todas, las cepas utilizadas para elaborar vino o cava. A pesar de que estos cambios son distinguibles por electroforesis por campo pulsante, uno de los retos claros en el desarrollo de las técnicas moleculares de identificación de levaduras es, sin duda alguna, el diseño de un procedimiento basado en PCR o en RFLP para detectar estos cambios, que pueden tener efectos muy perjudiciales en los procesos fermentativos y que, en todo caso, aportan una inseguridad indeseable.
Otras técnicas
Con el tiempo, se ha ido desarrollando una serie de técnicas de identificación de cepas de levaduras, que o bien no se utilizan tanto como las anteriores, o todavía resultan muy complicadas para su utilización sistemática. Aquí citaremos algunas:
Análisis de proteínas
El análisis de las proteínas de la levadura es un método mucho más sencillo que los métodos de identificación basados en la extracción del DNA. En cambio, es mucho menos sensible a variaciones genéticas entre cepas de levadura relativamente próximas. De todas formas, los avances de la proteómica y la electrofóresis de dos dimensiones podría volver a poner de moda este sistema en unos años.
Cromatografía de gases
Aunque se trate de una técnica complicada, el cromatógrafo de gases ya se ha convertido en un instrumento relativamente habitual en los laboratorios de enología. Por eso, los intentos de caracterizar cepas de levaduras por su perfil de ácidos grasos prometen ser muy interesantes, aunque la estandarización de los medios de cultivo y otros parámetros son críticos a la hora de asegurar la fiabilidad de los resultados.
RAPD
Esta PAPD (siglas que corresponden a Random Amplified Polymorphic DNA), o DNA polimórfico de amplificación aleatoria, es una técnica de análisis del DNA genómico por medio de PCR usando cebadores aleatorios. Tuvo una muy amplia aceptación hace algunos años, pero es difícilmente traspasable de un laboratorio a otro, dada su baja reproducibilidad.
Análisis de elementos de DNA repetitivo
A partir de los genes de los rRNA y del mitocondrial, el genoma de levadura tiene una serie de secuencias variables DNA (los loci ADH, MAL i SUC, las secuencias delta, o los elementos Ty o Y’ entre otros) que presentan cambios entre diferentes cepas, ya sea dentro de la propia secuencia, ya sea por su posición en el genoma. La posición de estas secuencias es muy variable, y en principio sería un buen método de identificación de cepas. Los métodos de detección de estas variaciones se basan normalmente en técnicas relativamente lentas y complicadas como la hibridación de DNA o de nested PCR (PCR anidado), que es un técnica mucho más reproducible que el RAPD, pero todavía relativamente complicada.
Análisis genómico
Las técnicas de biología molecular permiten observar la disposición de diversos elementos génicos con mucha precisión. De entrada, estas técnicas son lentas, complicadas y caras, por lo que no parecen ser las adecuadas para las necesidades industriales. No obstante, es previsible que la situación cambie radicalmente en unos (pocos) años: el análisis genómico (los famosos chips de DNA) pasarán a ser un protocolo rutinario en el ámbito clínico en pocos años, o quizá meses, y el sector agroalimentario le seguirá en poco tiempo. Preparar al sector para esta revolución es un reto que todos deberíamos proponer y compartir.
Bibliografía
Gasent-Ramírez J.M., Castrejón F., Querol A., Ramón D., Benítez T.: «Genomic stability of Saccharomyces cerevisiae baker’s yeasts», System Appl Microbiol 1999; 22: 329-340.
Nadal D., Colomer B., Piña B.: «Molecular polymorphism in phenotypically distinct populations of wine yeast strains», Appl Env Microbiol 1996; 62:1944-1950.
Nadal D., Carro D., Fernández-Larrea J., Piña B.: «Analysis and dynamics of the chromosomal complements of wild sparkling-wine yeast strains», Appl Env Microbiol 1999; 65: 1688-1695.
Naumov G.I.: «Genetic identification of biological species in the Saccharomyces sensu stricto complex», J Ind Microbiol 1996; 17: 295-302.
Pretorius I.S.: «Tailoring wine yeast for the new millenium: novel approaches to the ancient art of winemaking», Yeast 2000; 16: 675-729.
Querol A., Barrio E., Ramón D.: «A comparative study of different methods of yeast strain characterization», System Appl Microbiol 1992; 15: 439-446.





