Introducción
Los mapas genéticos permiten abordar el estudio del control genético de caracteres de interés
Al contrario de lo que ocurre en otros cultivos, en los que la innovación se fundamenta en el desarrollo de nuevas variedades, en viticultura y en enología la innovación se ha basado en la mejora de las técnicas agronómicas y en el desarrollo y aplicación de nuevas tecnologías en el proceso de fermentación. Así, mientras la mayoría de las variedades utilizadas en viticultura vienen multiplicándose vegetativamente durante siglos y, en algunos casos, su origen puede incluso trazarse hasta la época de los romanos (Mullins et al., 1992), ha sido la tecnificación de la viticultura y del proceso de fermentación los que han permitido la producción de vinos de gran calidad, con la necesaria reproducibilidad de cosecha en cosecha que el mercado demanda. Sin embargo, la innovación tecnológica en ambos procesos, viticultura y enología, no puede desarrollarse mucho más. La presión económica y social para cambiar los métodos productivos de la viticultura, en particular, y de la producción de frutales, en general, es cada vez mayor. Los costes de producción de la viticultura moderna intensiva son cada vez más elevados e incluyen un gran componente de coste energético. Concretamente, la protección química frente a las numerosas enfermedades de la vid no sólo es cara, sino que además provoca preocupaciones en los consumidores por los efectos secundarios en la salud y en el medio ambiente (Mullins et al., 1992; Bouquet et al., 2000). Toda esta situación nos lleva a la conclusión de que gran parte de los tratamientos químicos deben ser reemplazados por el uso de resistencias genéticas, y que una parte del coste de producción agronómica podría reducirse si se diera prioridad a la mejora genética. Innovar en viticultura en los próximos años puede depender más de la mejora genética que del desarrollo tecnológico, algo que, por otra parte, está por explotar en el mundo de la vid y el vino.
La mejora genética de especies leñosas presenta dificultades adicionales a la mejora de especie herbáceas. Estas dificultades aumentan en la uva de vinificación cuando se trata de mejorar variedades élite que representan genotipos únicos de gran calidad. La ingeniería genética es posiblemente la única estrategia que puede permitir la introducción de nuevos caracteres en estas variedades. Esta posibilidad exige un conocimiento amplio de la estructura y función del genoma de la vid y la identificación de las secuencias génicas que son responsables de los caracteres de interés. Este conocimiento permitirá también aumentar la eficacia de cualquier otra estrategia de mejora genética, al permitir la identificación y el seguimiento de las variantes génicas de interés.
En general, la mejora genética ha sido de menor aplicación en especies leñosas que en herbáceas dadas las características biológicas de las leñosas y, en particular, sus largos tiempos de generación. En la vid, el tiempo de generación, cuando se cultiva a partir de semilla, es de 4-5 años, lo que limita el número de generaciones que pueden llevarse a cabo en la vida profesional de un viticultor. Por otro lado, las variedades establecidas en vid son genotipos altamente heterocigóticos y los caracteres que definen su tipicidad segregan en la descendencia de los cruzamientos que se realicen con ellas. Como consecuencia, cualquier hibridación que realicemos con una variedad puede representar el inicio del desarrollo de nuevas variedades pero nunca permitirá la mejora de la variedad inicial para un nuevo carácter. Ambas características y el elevado conservadurismo del consumidor y del sector productivo han generado que la única mejora concebible en la vid de vinificación sea la selección clonal, o selección de aquellos clones resultado de la multiplicación vegetativa de una variedad que, como consecuencia de la mutación somática, muestran alguna nueva característica de interés. Desgraciadamente, el efecto negativo que tiene la hibridación en el desarrollo de nuevas variedades de vinificación se ha utilizado también para justificar el escaso interés de realizar cruzamientos entre distintas variedades de vid, visión que afortunadamente está quedando atrás, porque el análisis de la segregación de los caracteres en progenies derivadas de estos cruzamientos es la única estrategia para conocer la estructura genética de estos caracteres, y así ampliar el conocimiento genético de esta especie que, como comentamos al principio, es la única vía para la mejora genética de la especie.
En pocas palabras, si conocemos qué genes de la vid codifican distintas actividades enzimáticas como las de las rutas de síntesis de antocianos o de terpenos, y de qué manera contribuye cada uno al fenotipo final del color o el aroma del vino en las distintas variedades, se podrán diseñar estrategias muy eficientes de mejora genética de las variedades de vinificación. Éstas podrían abarcar, estrategias de selección clonal basadas en la búsqueda de variantes alélicas del gen de interés, estrategias de ingeniería genética mediante la transformación genética con variantes del gen de interés, o estrategias de mejora genética clásica centradas en la hibridación entre variedades y la selección de los híbridos portadores de las combinaciones génicas de interés. De igual manera podría hablarse de la resistencia a plagas y enfermedades o a condiciones ambientales extremas, que son las asignaturas pendientes en la mejora genética de la vid. Así, el conocimiento de la base genética de los caracteres de interés puede ser la base del desarrollo futuro de nuevas variedades y clones que, al igual que en otras especies, acabaran sustituyendo a las variedades actualmente en cultivo.
Desvelar la base genética de los caracteres de interés en una especie leñosa, y hacerlo con la precisión que requiere el análisis molecular, no es una tarea sencilla. Muchos de los caracteres de mayor interés, como la resistencia a patógenos, son caracteres cuantitativos, y su control genético viene determinado por múltiples loci con distintos efectos y con complejas interacciones genéticas y ambientales. Estos loci pueden ser, además, diferentes en distintas especies y variedades. Por ello, se hace necesaria como herramienta básica de trabajo la generación de múltiples poblaciones segregantes para los caracteres de interés y la construcción de mapas genéticos. Estos mapas son imprescindibles en el caso de caracteres cuantitativos y absolutamente necesarios cuando se quiere conocer la localización cromosómica y el efecto relativo de cada uno de los genes responsables de caracteres cualitativos. Afortunadamente, el desarrollo de la biología molecular en los últimos años ha generado múltiples herramientas moleculares o marcadores moleculares con los que saturar rápidamente un mapa genético. Además, el desarrollo de la genética en especies no mejoradas permite contar con nuevos métodos de mapeo para la construcción de mapas genéticos en estas especies.
Herramientas moleculares empleadas en la construcción de mapas genéticos
Para abordar la construcción de los mapas genéticos disponemos de numerosas clases de marcadores moleculares, que se diferencian en su carácter dominante o codominante (Karp y Edwards, 1998). Los marcadores dominantes, como los RAPD1 y los marcadores de alta eficacia (AFLP2 y SAMPL3), permiten el análisis de un elevado número de loci por experimento sin requerir información previa sobre su secuencia, empleándose en la construcción del armazón de los mapas genéticos en el que se localizan los marcadores codominantes. Por otro lado, los marcadores codominantes como RFLP4, microsatélites (SSR5), STS6, CAP7, EST8 y SNP9, aunque permiten analizar sólo un locus por experimento, son más informativos al permitir diferenciar las variantes alélicas de los loci analizados y por lo tanto identificar grupos de ligamiento entre diferentes mapas genéticos. Sin embargo, para su desarrollo se precisa conocer la secuencia y, por lo tanto, son menos numerosos que los dominantes.
Estrategias de mapeo en especies no mejoradas
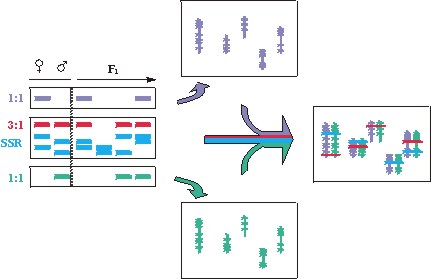
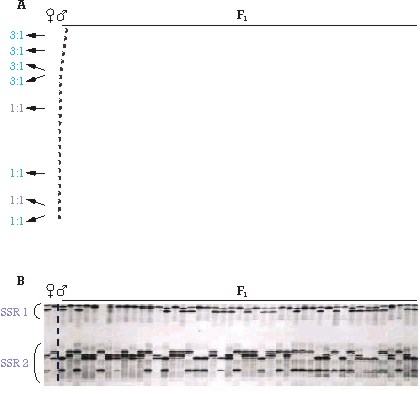
Tradicionalmente los mapas genéticos se han desarrollado a partir del estudio de segregación de marcadores en poblaciones derivadas de cruces entre líneas puras, progenies F2, o retrocruzamientos, pedigríes de los que no se dispone, en la actualidad, en especies leñosas (forestales y árboles frutales). En 1994 Gratapaglia y Sederoff publicaron los primeros mapas de eucaliptos basados en la combinación de una nueva estrategia de mapeo, el pseudocruzamiento prueba o pseudoretrocruzamiento en dos sentidos, con marcadores dominantes (RAPD1). Esta estrategia aprovecha el elevado nivel de heterocigosidad que muestran las especies no mejoradas, como por ejemplo la vid, para construir mapas genéticos estudiando la segregación de marcadores en progenies F1, es decir las derivadas del cruce controlado entre dos plantas. Se denomina pseudocruzamiento prueba «en dos sentidos» porque permite obtener de forma simultánea los mapas genéticos de ambos progenitores, empleando marcadores que segregan 1:1 en la progenie, es decir, presentes en heterocigosis en uno de los progenitores y ausentes en el otro (figura 1). La utilización de marcadores dominantes de alta eficacia, como AFLP y SAMPL (figura 2), constituye la herramienta clave para construir, de forma rápida, la estructura de los mapas genéticos, así como saturar regiones genómicas de interés. Los marcadores dominantes heterocigotos en ambos progenitores (que muestran segregación 3:1) y los marcadores codominantes, como microsatélites (SSR, figura 2), permiten identificar grupos de ligamiento homólogos entre los mapas genéticos de los progenitores (figura 1). Los marcadores codominantes, como se indicó previamente, también se emplean para la identificación de grupos de ligamiento homólogos entre mapas genéticos desarrollados por diferentes grupos de investigación y, de esta manera, comparar los resultados del análisis de caracteres.
Mapas genéticos de vid
Los primeros mapas genéticos del género Vitis fueron desarrollados por Lodhi et al. (1994, 1995a, 1995b), quienes elaboraron los mapas de ligamiento de los híbridos inter-específicos ‘Cayuga White’ (híbrido complejo de V. vinifera, V. labrusca, V. rupestris y V. aestivalis) y ‘Aurore’ (híbrido complejo de V. vinifera, V. rupestris y V. aestivalis), mediante el estudio de una pequeña progenie F1, compuesta por tan sólo 60 individuos, con 15 isoenzimas, 13 RFLP y 422 RAPD. Estos mapas se están empleando en el estudio de caracteres relacionados con resistencias a oídio y podredumbre gris, tolerancia a frío, y con producción (tamaño y número de racimos y bayas, y tiempo de floración y maduración). Este grupo de investigación también elaboró los mapas genéticos de ‘Horizon’ (Seyval x Schuyler) e ‘Illinois 547-1’ (V. cinerea B9 x V. rupestris B38) estudiando la segregación de 277 RAPD, 25 microsatélites, 4 CAP y 12 AFLP en 58 individuos progenie resultantes del cruce inter-específico entre ambos clones (Dalbó et al., 2000b). En el mapa genético de ‘Illinois 547-1’ se ha localizado un locus único que determina que sus flores sean masculinas, identificándose dos marcadores ligados a dicho locus. Estos mapas también se están empleado en el análisis del control genético de la resistencia cuantitativa a oidio y a podredumbre negra y producción de resveratrol (Dalbó et al., 1997, 2000a). Simultáneamente, se han identificado grupos de ligamiento homólogos entre los cuatro mapas genéticos obtenidos por este grupo de investigación, con el fin de analizar la capacidad de transferencia de marcadores entre dichos mapas, y la estabilidad de los loci detectados, responsables de los caracteres de interés.
En la actualidad se están desarrollando proyectos centrados en el estudio de caracteres de interés, tanto de variedades de vinificación como de uva de mesa, por lo que se están generando diversos mapas genéticos. Los alemanes Buck y Zyprian (2000) están construyendo, con RAPD y SCAR10, los mapas genéticos de las variedades de V. vinifera ‘Regent’ y ‘Lemberger’, resistente y susceptible a mildiu, oidio y podredumbre gris, respectivamente, con objeto de estudiar la base genética de dichas resistencias. El grupo italiano integrado por Grando et al. (2000) está construyendo los mapas genéticos de V. vinifera cv. moscato bianco y V. riparia con AFLP2 y microsatélites, para identificar en el mapa de V. riparia los loci que controlan la resistencia a mildiu y oidio. El mapa resultante del análisis de la segregación de microsatélites en la progenie obtenida mediante cruzamiento controlado entre V. vinifera cv. riesling y V. vinifera cv. cabernet sauvignon se está empleando para estudiar caracteres de interés en vinificación relacionados con la producción y calidad, como color y peso de la baya, concentración de antocianos en la baya y peso del racimo, identificando un locus que controla el color del hollejo (Riaz y Meredith, 2000). Algunas bodegas como E. & J. Gallo Winery y Sun World International, Inc. también están desarrollando mapas genéticos de híbridos, empleando AFLP y microsatélites, para conocer el control genético de diferentes caracteres de interés (Georgiady et al., 2001).
Uno de los caracteres de mayor interés en la mejora de uva de mesa es la apirenia o falta de semilla. Con objeto de estudiar el control de la apirenia y otros caracteres como los relacionados con baya, racimo y tiempo de maduración, se están elaborando diferentes proyectos de mapeo. En Francia, Doligez et al. (2000) están abordando este análisis mediante la construcción de los mapas genéticos de dos genotipos parcialmente apirenos: ‘2223-27’ (Alfonso Lavallée x Sultanina) y ‘2121-30’ (Dattier Beyrouth x 75 Pirovano), empleando mayoritariamente AFLP, microsatélites, isoenzimas y un SCAR asociado a la apirenia (Lahogue et al., 1998). Nuestro grupo de investigación está generando los mapas genéticos de dos variedades de vid, una pirena local ‘Dominga’ y una apirena ‘Autum Seedless’, mediante el estudio de segregación de marcadores de alta eficacia (AFLP, SAMPL) y microsatélites (Cabezas et al., 2001). Estos mapas permitirán estudiar, además de la apirenia, otros caracteres como fotorreacción (acumulación de antocianos en el hollejo provocado por la radiación solar), tiempo de floración, tiempo de maduración y vigor de la cepa.
Análisis de QTL y mejora molecular
Las regiones del genoma que controlan caracteres cuantitativos (QTL, de Quantitative Trait Loci) como resistencias a plagas y enfermedades, tolerancia a condiciones ambientales extremas, producción (número y tamaño de bayas y racimos, compactación, precocidad, porte de la planta) y calidad (color, sabor amoscatelado, textura) se estudian buscando asociaciones entre marcadores integrados en los mapas genéticos y el fenotipo segregante de la progenie empleada en el mapeo. La identificación y utilización de estos marcadores asociados a caracteres de interés agronómico permitirá, en la mejora de uva de mesa, la evaluación precoz y selección de individuos progenie portadores del carácter deseado. En el caso de variedades de vinificación, estos marcadores permitirán seleccionar los genotipos con el o los caracteres deseados y concentrar sobre ellos el trabajo de selección clonal. Por otro lado, las regiones del genoma donde se localizan los loci responsables de dichos caracteres y los marcadores asociados a los mismos, constituyen el punto de partida para iniciar el clonaje posicional de genes que controlan estos caracteres, mediante saturación de la región que contiene el gen con marcadores flanqueantes (Tanksley, 1995). Una vez identificados y caracterizados, estos genes pueden emplearse en estrategias de ingeniería genética para introducir dichos caracteres en variedades muy heterocigotas, como son las de vid, y mantener la combinación génica de los genotipos de interés.
Agradecimientos
Este trabajo comenta algunos de los resultados del desarrollo de mapas genéticos de vid mediante AFLP, SAMPL y microsatélites realizados en colaboración con José Luis Cénis y Juan Carreño, del CIDA (Murcia). Nuestra investigación en vid está financiada por el proyecto COO1999A X087.
Notas
1 RAPD: Random Amplified Polymorphic DNA (DNA polimórfico amplificado al azar).
2 AFLP: Amplified Fragment Length Polymorphism (polimorfismo de la longitud de los fragmentos amplificados).
3 SAMPL: Selective Amplification of Microsatellite Polymorphic Loci (amplificación selectiva de loci polimórficos de microsatélites).
4 RFLP: Restriction Fragment Length Polymorphism (polimorfismo en el tamaño de los fragmentos de restricción).
5 SSR: Simple Sequence Repeat (repetición de secuencias discretas).
6 STS: Sequence Tagged Site (lugar etiquetado para la secuencia).
7 CAPS: Cut Amplified Polymorphic Sequence (secuencia polimórfica amplificada y cortada).
8 EST: Expressed Sequence Tag (lugar etiquetado para la expresión).
9 SNP: Single Nucleotide Polymorphism (polimorfismo en un único nucleótido).
10 SCAR: Sequence Characterized Amplified Region (región amplificada caracterizada y secuenciada).
Bibliografia
Bouquet, A., Torregrosa, L., Danglot, Y., Bongiovanni, M., Castagnone-Sereno, P., Esmenjaud, D., Dalmsso, A.: «Breeding rootstocks resistant to grape fanleaf virus spread, using Vitis x Muscadinea hybridization », Proceedings of the Seventh International Symposium on Grapevine Genetics and Breeding, Acta Horticultura 2000: 203-207.
Buck, S., Zyprian, E.: «First approaches of molecular mapping in a model population derived from the crossing of the grapevine varieties ‘Regent’ x ‘Lemberger’», Proceedings of the Seventh International Symposium on Grapevine Genetics and Breeding, Acta Horticultura 2000: 203-207.
Cabezas, J.A., Cervera, M.T., Cenís, J.L., Zapater, J.M.: «Construcción de un mapa genético de AFLPs y microsatélites en Vitis vinifera», IV Reunión de Biología Molecular de Plantas, 2001, Toledo.
Dalbó M.A., Ye G.-N., Weeden N.F., Reisch B.I.: «QTL analysis of powdery mildew resistance in grapes», Plant & Animal Genome Conference V, San Diego, 1997.
Dalbó M.A., Weeden N.F., Reisch B.I.: «QTL analysis of disease resistance in interspecific hybrid grapes», Proceedings of the Seventh International Symposium on Grapevine Genetics and Breeding, Acta Horticultura 2000a: 215-219.
Dalbó M.A., Ye G.-N., Weeden N.F., Steinkellner H., Reisch B.I.: «A gene controling sex in grapevines placed on a molecular marker-based genetic map», Genome 2000b; 43: 333-340.
Doligez A., Adam-Blondon A.-F., Ballaquestr J.-F., Bouquet A., Roux C., This P.: «Construction of a Vitis vinifera linkage map for the detection of QTLs of several bunch quality traits», Plant & Animal Genome Conference VIII, San Diego, 2000.
Georgiady M., DeScenzo R., Cain D.W., Irelan N.: «Linkage mapping and QTL mapping in grape», Plant & Animal Genome Conference IX, San Diego, 2001.
Grando M.S., Bellin D., Madini A., Stefanini M., Pozzi C., Velasco R.: «Construction of an AFLP and SSR genetic map of Vitis from an interspecific hybrid population», Plant & Animal Genome Conference VIII, San Diego, 2000.
Grattapaglia D., Sederoff R.: «Genetic linkage maps of Eucalyptus grandis and Eucalyptus urophylla using a pseudo-testcross: mapping strategy and RAPD markers», Genetics 1994; 137: 1121-1137.
Karp A., Edwards K.J.: «DNA markers: a global overview», en: DNA markers: protocols, aplications and overviews. Eds: G. Caetano- Anollés i P.M. Gresshoff, Wiley, New York, 1998: 1-13.
Lahogue F., This P., Bouquet A.: «Identification of a codominant SCAR marker linked to the seedlessness character in grapevine», Theoretical and Applied Genetics 1998; 97: 950-959.
Lodi M.A., Reisch B.I., Bruce I., Weeden N.F.: «Genetic linkage maps of Vitis and QTL detection by interval mapping», Plant Genome Conference II, San Diego, 1994.
Lodi M.A., Daly M.J., Ye G.-N., Weeden N.F., Reisch B.I.: «A molecular marker linkage map of Vitis», Genome 1995a; 38: 786-794.
Lodi M.A., Moreno S., Weeden N.F., Bruce I., Reisch B.I.: «Detection of Quantitative Trait Loci controlling important traits and characterization of RAPD markers in Vitis», Plant Genome Conference III, San Diego, 1995b.
Mullins M.G., Bouquet A., Williams L.E.: «Biology of the grapevine», Cambridge, Gran Bretaña, Cambridge University Press, 1992.
Riaz S., Meredith C.: «A microsatellite marker based linkage map of Vitis vinifera», Plant & Animal Genome Conference VIII, San Diego, 2000.
Tanksley S.D., Ganal M.W., Martin G.B.: «Chromosome landing: a paradigm for map-based gene cloning in plants with large genomes», Trends Genet 1995; 11: 63-68.