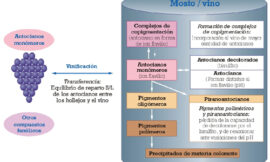
Las aminas biógenas (AB) son bases orgánicas de bajo peso molecular, tienen al menos un grupo amino en su estructura y carácter básico conferido por este grupo amino. Pueden estar presentes de forma natural en alimentos fermentados o pueden ser formadas por microorganismos como consecuencia de la descarboxilación de los aminoácidos presentes en esos alimentos. Las AB tienen efectos toxicológicos cuando se encuentran en alta concentración porque pueden producir efectos fisiológicos indeseables en el consumidor como náuseas, dolores de cabeza, alteraciones respiratorias, enrojecimiento de la piel, palpitaciones cardíacas, hiper- o hipotensión y fenómenos de alergia. La mayoría de las intoxicaciones alimentarias se relacionan fundamentalmente con la histamina y tiramina. El resto de las AB se considera que pueden contribuir a reforzar la acción de las anteriores.1
La síntesis de AB supone la coincidencia de tres factores diferentes: existencia de precursores (aminoácidos), presencia de microorganismos con actividades descarboxilásicas correspondientes, y concurrencia de las condiciones ambientales adecuadas.2 La existencia de aminoácidos se encuentra cualitativa y cuantitativamente ligada a las materias primas y a la tecnología de vinificación: variedad de uva, momento de la vendimia, adición de nutrientes aminoacídicos, maceración, adición de enzimas, lisis microbiana, etc.2 La existencia de condiciones favorables para el metabolismo microbiano, como por ejemplo valores de pH elevados, bajo contenido en SO2, etc. favorecerán lógicamente la síntesis de AB.
Origen de las aminas biógenas
Las bacterias lácticas (BL) son las principales responsables de la síntesis de AB en vino.3 En la tabla 1 puede observarse la correlación entre la especie de BL y el tipo de AB sintetizada en vino.3 En el caso de la histamina, la producción de este compuesto es un carácter dependiente de cepa, variando desde un 6% aproximadamente de las cepas positivas para la especie L. mesenteroides, a un 80% de positivas en la especie O. oeni.1,4-14 Hay que señalar que estos porcentajes varían en función del método empleado, ya que los sistemas menos sensibles como la detección en placa solamente revelarán las cepas que produzcan concentraciones más elevadas de AB, mientras que métodos enzimáticos o que emplean HPLC serán capaces de mostrar cepas que produzcan concentraciones más bajas de AB.9-15 En cuanto a la tiramina, en algunas especies como L. brevis, todas las cepas analizadas aisladas de vino son productoras, mientras que por ejemplo sólo una parte de las cepas pertenecientes a L. hilgardii y L. plantarum sintetizan esa AB.16,17 Es de destacar que todas las cepas productoras de tiramina son capaces de sintetizar feniletilamina, quizá debido a una actividad secundaria del mismo enzima descarboxilásico.17 Las especies de BL aisladas de vinos que son capaces de sintetizar putrescina son L. buchneri,15 L. hilgardii,11 O. oeni 7 y quizá L. brevis.18 Este carácter parece ser más escaso en BL que la capacidad para sintetizar otras AB. De todas formas, el número de cepas capaces de sintetizar putrescina parece que es cada vez más frecuente: hace algunos años no se aislaban BL del vino productoras de putrescina, y cada vez parece que la frecuencia de su aislamiento es mayor, lo cual podría significar a su vez que únicamente se han mejorado los métodos de detección y cuantificación de esas AB.
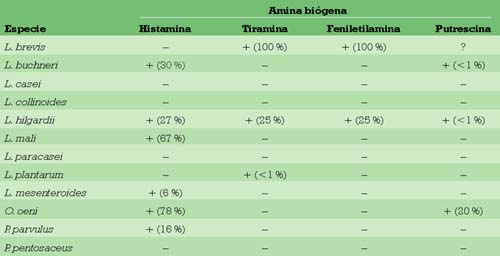
∗ El símbolo + significa que el microorganismo es productor de la amina, el – que no lo es, el ? que la actividad es putativa, todavía no demostrada, y el número que aparece entre paréntesis indica el porcentaje de microorganismos que hemos encontrado que son productores de cada AB
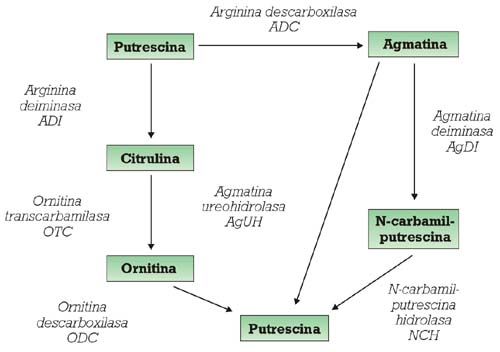
Las AB son producidas a partir de los aminoácidos precursores gracias a la acción de las descarboxilasas correspondientes. Así, la histidina descarboxilasa (HDC) sintetiza histamina a partir de histidina, la tiramina descarboxilasa (TDC) sintetiza tiramina desde tirosina y feniletilamina a partir de fenilalanina. El caso de la putrescina es más complejo (fig. 1). La ruta de la arginina deiminasa está asociada a la especie de O. oeni, 7 y la ruta de la agmatina a L. hilgardii.19
Detección de aminas biógenas
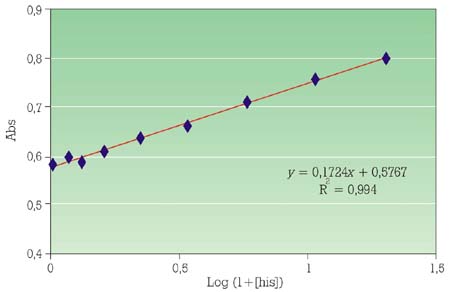
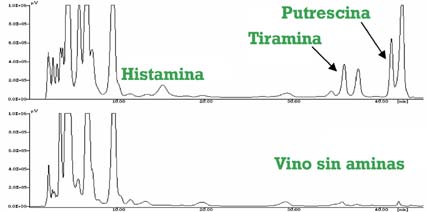
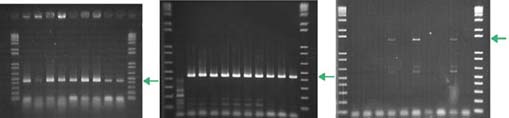
Debemos tener en cuenta que existen múltiples métodos para la detección y cuantificación de aminas biógenas y para la identificación de los microorganismos responsables de dicha producción. Podemos encontrar desde métodos en placa (fig. 2),3 métodos inmunológicos (ELISA),20métodos enzimáticos (fig. 3)15 hasta métodos por HPLC (fig. 4).3 Cada método presenta sus ventajas e inconvenientes, incluyendo precio, rapidez, fiabilidad, dificultad, necesidad de personal entrenado y equipamiento, etc. De ahí se deriva en parte el que existan resultados divergentes entre diferentes laboratorios e investigadores, lo que nos lleva a la necesidad de estandarizar este tipo de análisis. Lo deseable sería acordar un método oficial de análisis de AB, o al menos de referencia, no sólo entre la comunidad investigadora sino más aún en el sector de producción industrial.
En cuanto a la identificación de los microorganismos causantes de la síntesis de AB en vinos, se está utilizando la PCR de genes simple o múltiple. Hoy en día existen cebadores específicos para detectar los genes correspondientes a los enzimas descarboxilásicos (hdc, tdc y odc). En el caso del gen tdc, observamos un ejemplo en la figura 5a,17,21para el caso del gen hdc se muestra en la figura 5B.9 En cuanto a la síntesis de putrescina, detectamos el gen odc (fig. 5C). 4,13,22,23 En los tres casos consideramos positivas aquellas reacciones de PCR que den una banda específica a la altura correspondiente al tamaño esperado del gen amplificado.
Al igual que con la detección de la producción de AB, existen varios sistemas de PCR que permiten detectar la presencia de los genes responsables de dicha síntesis, pero no en todos los casos se produce la reacción específica debida. En algunas ocasiones, se pueden llegar a dar falsos positivos o falsos negativos.24
El protocolo en el laboratorio Para la realización de todas estas pruebas debemos, en primer lugar, aislar los microorganismos presentes en la muestra, utilizando placas de cultivo de diferentes medios (MRS, MLO…), realizando diluciones seriadas o filtrando un volumen conocido de la muestra. Las placas se dejan incubar en una estufa a 28 ºC durante 4-7 días. Tras estos días y observando crecimiento de colonias realizamos un recuento, siembra en triple estría para purificar y posteriormente liofilizar las cepas para su conservación, así como todas las pruebas de PCR antes mencionadas. A continuación todas las bacterias lácticas aisladas y que han dado positivas en la amplificación de los genes y en la producción de AB (por HPLC y método enzimático), se reinoculan por separado en vino para corroborar la capacidad de producción de aminas en vino. Este es un punto clave. Si realmente queremos conocer la capacidad de una determinada cepa de BL para alterar un vino produciendo AB, hemos de resembrarla en este tipo de vino y observar si puede sintetizar esas AB de nuevo. Como hemos dicho, según las condiciones del vino puede haber mayor o menor facilidad para sintetizar una determinada AB. De esta forma, averiguaremos la peligrosidad real de un determinado microorganismo, o si la alteración detectada en un vino se debe realmente a una bacteria o a otra. Como ejemplo, ya hemos citado que alrededor el 80% de las cepas de O. oeni pueden sintetizar histamina, pero realmente en condiciones reales de vinificación suelen contribuir menos que otras bacterias, y son cepas de Pediococcus parvulus o Lactobacillus hilgardii las que realmente suelen producir las mayores cantidades de histamina en vinos.9,25 |
Conclusión
Para concluir debemos tener en cuenta las limitaciones de cada método de cuantificación de AB o de detección de los genes responsables de la síntesis de estos compuestos. Es el caso del método de detección de bacterias productoras de AB en placa, ya que es un sistema menos sensible y por lo tanto detectará solamente las cepas con mayor capacidad de producción, y sin poder además cuantificar dichas AB. Tendremos que ser cuidadosos también en el manejo de las técnicas moleculares, ya que un error en la pareja de cebadores elegidos nos estaría dando falsos negativos en la amplificación de los genes. Por todo ello, debemos ser cautos a la hora de comparar resultados y sacar conclusiones. Consideramos totalmente necesaria y urgente la normalización de métodos, y por lo tanto se debería establecer algún método oficial para el análisis de aminas biógenas y de los genes implicados, o al menos algún método de referencia. Pensamos que también es necesario determinar unos límites máximos de cada AB en vino, ya que por el momento sólo encontramos recomendaciones o límites a la importación en algunos países.
Agradecimientos
Este trabajo ha sido financiado por los proyectos AGL2000-0827-CO2-01, VIN03-014-C3-1 y AGL2006-08495/ALI.
Bibliografía
- Landete, J.M.; Pardo, I.; Ferrer, S.: «Histamine, histidine, and growth-phase mediated regulation of the histidine decarboxylase gene in lactic acid bacteria isolated from wine», FEMS Microbiology Letters 2006; 260: 84-90.
- Ferrer, S.; Pardo, I.: «Prevención de la aparición de aminas biógenas en vinos» ACE Revista de Enología 2005; 70: 6-8.
- Landete, J.M.: «Estudio y caracterización molecular de la producción de aminas biógenas por parte de bacterias lácticas de origen enológico», Tesis Doctoral. Universitat de València, 2005.
- Coton, E.; Coton, M.: «Multiplex PCR for colony direct detection of Gram-positive histamine- and tyramine-producing bacteria», Journal of Microbiological Methods 2005; 63: 296-304.
- Coton, E.; Rollan, G.C.; Lonvaud-Funel, A.: «Histidine carboxylase of Leuconostoc oenos 9204: purification, kinetic properties, cloning and nucleotide sequence of the hdc gene», J Appl Microbiol 1998; 84: 143-151.
- De las Rivas, B.; Marcobal, Á.; Muñoz, R: «Improved multiplex-PCR method for the simultaneous detection of food bacteria producing biogenic amines», FEMS Microbiology Letters 2005; 244: 367-372.
- Guerrini, S.; Mangani, S.; Granchi, L.; Vincenzini, M.: «Biogenic amine production by Oenococcus oeni», <em 2002; 44: 374-378.</em
- Landete, J.M.; Ferrer, S.; Pardo, I: «Biogenic amine production by lactic acid bacteria, acetic bacteria and yeast isolated from wine», Food Control 2007; 18: 1569-1574.
- Landete, J.M.; Ferrer, S.; Pardo, I.: «Which lactic acid bacteria are responsible of histamine production in wine?», Journal of Applied Microbiology 99: 580-586.
- Lonvaud-Funel, A.: «Biogenic amines in wines: role of lactic acid bacteria», FEMS Microbiology Letters 2001; 199: 9-13.
- Lonvaud-Funel, A.; Joyeux, A.: «Histamine production by wine lactic acid bacteria: isolation of a histamine-producing strain of Leuconostoc oenos», Journal of Applied Bacteriology 1994; 77: 401-407.
- Lucas, P.M.; Wolken, W.A.M.; Claisse, O.; Lolkema, J.S.; Lonvaud-Funel, A.: «Histamine-Producing Pathway Encoded on an Unstable Plasmid in Lactobacillus hilgardii 0006», Appl. Environ. Microbiol. 2005; 71: 1417-1424.
- Marcobal, Á.; Rivas, B.; Moreno-Arribas, M.V.; Muñoz, R.: «Multiplex PCR method for the simultaneous detection of histamine-, tyramine-, and putrescine-producing lactic acid bacteria in foods», Journal of Food Protection 2005; 68: 874-878.
- Moreno-Arribas, M.V.; Polo, M.C.; Jorganes, F., Muñoz, R.: «Screening of biogenic amine production by lactic acid bacteria isolated from grape must and wine», International Journal of Food Microbiology 2003; 84: 117-123.
- Landete, J.M.; Ferrer, S.; Pardo, I.: «Improved enzymatic method for the rapid determination of histamine in wine», Food Additives and Contaminants 2004; 21: 1149-1154.
- Arena, M.; Fiocco, D.; Manca de Nadra, M.; Pardo, I.; Spano, G.: «Characterization of a Lactobacillus plantarum Strain Able to Produce Tyramine and Partial Cloning of a Putative Tyrosine Decarboxylase Gene», Current Microbiology 2007; 55: 205-210.
- Landete, J.M.; Pardo, I.; Ferrer, S.: «Tyramine and phenylethylamine production among lactic acid bacteria isolated from wine», International Journal of Food Microbiology 2007; 115: 364-368.
- Lucas, P.M.; Blancato, V.S.; Claisse, O.; Magni, C.; Lolkema, J.S.; Lonvaud-Funel, A.: «Agmatine deiminase pathway genes in Lactobacillus brevis are linked to the tyrosine decarboxylation operon in a putative acid resistance locus», Microbiology2007; 153: 2221-2230.
- Arena, M.E.; Landete, J.M.; Manca de Nadra, M.C.; Pardo, I.; Ferrer, S.: «Factors affecting the production of putrescine from agmatine by Lactobacillus hilgardii X1B isolated from wine», Journal of Applied Microbiology 2008; 105: 158-165.
- Marcobal, A.; Polo, M.C.; Martín-Álvarez, P.J.; Moreno-Arribas, M.V.: «Biogenic amine content of red Spanish wines: comparison of a direct ELISA and an HPLC method for the determination of histamine in wines», Food Research International 2005; 38: 387-394.
- Lucas, P.; Lonvaud-Funel, A.: «Purification and partial gene sequence of the tyrosine decarboxylase of Lactobacillus brevis IOEB 9809», FEMS Microbiology Letters 2002; 211: 85-89.
- Landete, J.M.; Arena, M.E.; Pardo, I.; Manca de Nadra, M.; Ferrer, S.: «Implicated bacterial genes in putrescine production from agmatine: detection and correlation with the activity», FEMS Microbiology Letters 2007 (enviat).
- Marcobal, A.; De las Rivas, B.; Moreno-Arribas, M.V.; Muñoz, R.: «Identification of the ornithine decarboxylase gene in the putrescine-producer Oenococcus oeni BIFI-83», FEMS Microbiology Letters 2004; 239: 213-220.
- Ferrer, S.; Landete, J.M.; Polo, L.; Pardo, I.: «Las bacterias y su repercusión sobre las aminas biógenas», Le Bulletin de l’OIV 2007; 80: 293-302.
- 25. Landete, J.M.; Ferrer, S.; Polo, L.; Pardo, I.: «Biogenic amines in wines from three Spanish regions», Journal of Agricultural and Food Chemistry 2005; 53: 1119-1124.