 Aparte de animales y plantas, los humanos también hemos ido “domesticando” algunos microbios a lo largo de la historia, al ir desarrollando los alimentos fermentados. Los grupos principales de estos microorganismos son las bacterias lácticas de muchos productos lácteos y vegetales fermentados, las levaduras de las bebidas alcohólicas y los mohos de algunos quesos y derivados de la soja. En este artículo, después de una breve introducción, nos limitaremos a los microbios del vino, o sea, a las levaduras de la fermentación alcohólica como Saccharomyces cerevisiae y a las bacterias de la fermentación maloláctica como Oenococcus oeni.
Aparte de animales y plantas, los humanos también hemos ido “domesticando” algunos microbios a lo largo de la historia, al ir desarrollando los alimentos fermentados. Los grupos principales de estos microorganismos son las bacterias lácticas de muchos productos lácteos y vegetales fermentados, las levaduras de las bebidas alcohólicas y los mohos de algunos quesos y derivados de la soja. En este artículo, después de una breve introducción, nos limitaremos a los microbios del vino, o sea, a las levaduras de la fermentación alcohólica como Saccharomyces cerevisiae y a las bacterias de la fermentación maloláctica como Oenococcus oeni.
Domesticación
Cuando el flujo genético entre poblaciones de la misma especie se reduce, usualmente por aislamiento, la evolución puede dar lugar a subpoblaciones diferentes, que eventualmente pueden convertirse en especies diferentes. Los humanos pueden interferir en este proceso natural limitando el flujo genético deliberadamente, seleccionando determinados rasgos deseados, o sea, la selección artificial. De esta forma, algunas especies acaban siendo dependientes de ambientes antropogénicos y adquieren rasgos evolutivos que interesan a los humanos. Esto es la domesticación.
Los rasgos buscados son muy diversos, desde el rendimiento (ej. ganadería bovina u ovina o la agricultura intensiva), pasando por la tolerancia al estrés (ej. cosechas resistentes a la sequía), hasta llegar a la estética (ej. flores) o a la compañía (ej. perros). Con la excepción de los perros, ya domesticados hace unos 30 000 años, la mayoría de especies domesticadas lo fueron durante la revolución neolítica hace unos 12 000 años, con el cambio del nomadismo de cazar y recolectar al sedentarismo ligado a los cultivos y al ganado. Al seleccionar los descendientes de animales y plantas que mejor cubrían las necesidades específicas, estos humanos prehistóricos efectivamente empezaron a controlar los procesos de selección y el flujo genético (Steensels et al., 2019).
Domesticación de microorganismos
Comparado con la domesticación de plantas y animales, la de microbios ha sido ignorada mucho tiempo. Recientemente, el gran aumento de datos genómicos disponibles ha permitido conocer con precisión las relaciones genéticas entre distintos microorganismos, con lo que se ha visto que los microbios industriales a menudo representan ramas genéticas diferenciadas en los árboles filogenéticos. Sin embargo, las circunstancias ambientales que requiere la domesticación microbiana y las características genéticas concretas no se han estudiado en detalle.
Curiosamente, mientras que la domesticación de animales y plantas son sobre todo el resultado del esfuerzo humano, la domesticación de los microbios ha sucedido mayoritariamente sin intención. Cabe recordar que la existencia de los microorganismos empezó a finales del s. XVII con Antonvan Leeuwenhoek, su papel en las fermentaciones no fue desvelado hasta el s. XIX con Louis Pasteur y otros, y sobre todo los primeros cultivos puros, en concreto de levaduras cerveceras, no fueron obtenidos hasta finales del s. XIX por Emil C. Hansen en la cervecería Carlsberg.
Sin embargo, las fermentaciones son conocidas desde el neolítico o antes, aunque no se supiera nada de los microbios. Uno de los primeros pasos para controlar un poco el proceso de fermentación fue probablemente la retro-inoculación (backslopping en inglés), donde se utiliza material —y por tanto microbios— de una fermentación previa para empezar una nueva. Con esto, aquellos antiguos artesanos promovieron sin pretenderlo la adaptación de los microbios al ambiente fermentativo antropogénico, fue una selección “inconsciente”.
Cuando el flujo genético entre poblaciones de la misma especie se reduce, usualmente por aislamiento, la evolución puede dar lugar a subpoblaciones diferentes, que eventualmente pueden convertirse en especies diferentes. Los humanos pueden interferir en este proceso natural limitando el flujo genético deliberadamente, seleccionando determinados rasgos deseados, o sea, la selección artificial.
Durante el proceso de domesticación de los microbios, existe una competencia continua entre diferentes variantes, ya sean especies, cepas o mutantes, en cada ronda de fermentación. O sea, también existe una supervivencia de los más adaptados, como en la selección natural, aunque aquí están en un ambiente controlado por los humanos. Sin embargo, esto ha cambiado en estos últimos decenios desde que se utiliza la inoculación de cultivos puros, seleccionados previamente en base a las características buscadas. Esta selección más “artificial” se asemeja más a la domesticación tradicional de animales y plantas. La introducción de las nuevas herramientas biotecnológicas en los últimos años ha permitido diseñar aún mejor el microorganismo con las propiedades deseadas para la fermentación, incluyendo algunas, como la producción de aromas exóticos, que no necesariamente dan una ventaja adaptativa al microorganismo.
Recomiendo la revisión de Steensels et al. (2019) para repasar las características del proceso de domesticación en los principales grupos de microbios industriales, qué son las bacterias lácticas, levaduras y mohos. Como he mencionado anteriormente, aquí me centraré sobre todo en los relacionados con el proceso de vinificación, o sea levaduras y bacterias lácticas, y por este orden de relevancia y temporalidad en el vino.
Domesticación de las levaduras
Las levaduras son hongos unicelulares, y por tanto microscópicos. Tienen unos 4 micrómetros de diámetro, aunque algunos pueden llegar hasta 40 micrómetros, y ocasionalmente algunas hacen prolongaciones o cadenas de células conocidas como pseudohifas. Se reproducen sobre todo asexualmente por mitosis con gemación, una división asimétrica donde la levadura menor es la gema. En condiciones de estrés pueden esporular y después de la meiosis se producen esporas haploides que pueden conjugarse. La mayoría son ascomicetos pero también las hay de basidiomicetos, y aunque hay unas 1500 especies de levaduras, la “levadura” por antonomasia es Saccharomyces cerevisiae.
Aunque no exclusivas del vino, las levaduras S. cerevisiae son el modelo perfecto de domesticación microbiana porque, pese a ser eucariotas, tienen un genoma pequeño (12 Mb y 6500 genes, frente a la media en los ascomicetos que es de 36 Mb y 11 000 genes) muy estudiado, tienen tiempos de generación cortos y se dispone de muchas herramientas para realizar investigación genética y fenotípica con ellas.
S. cerevisiae solo es abundante en las fermentaciones de fruta recogida artificialmente, como en las uvas vendimiadas una vez están en la bodega. En cambio, de forma natural se han encontrado en muchos hábitats pero en números escasos, y en particular en las frutas, en la uva misma, y en la corteza de los robles, que parecen ser sus nichos ecológicos, pero en cualquier caso en números escasos (Steensels et al., 2019).
Por eso se ha propuesto que S. cerevisiae no es que esté adaptada a ningún nicho específico sino que puede decirse que es una especie nómada, capaz de sobrevivir como generalista poco abundante en un amplio abanico de ambientes. Puede sobrevivir en muy diversas condiciones y aunque su genoma es pequeño, es bastante complejo y de hecho para crecer en los medios de laboratorio solo necesita el 20% de sus 6500 genes (Goddard &Greig, 2015).
Pero a pesar de no tener claro cuál es su hábitat natural, está claro que su segundo hábitat son muchos ambientes artificiales de fermentaciones como las de pan, cerveza y vino. S. cerevisiae es conocido por sus excelentes características fermentativas, incluso en presencia de oxígeno. Este carácter surgió como un proceso evolutivo, adquiriendo la capacidad de crecer en medios con muchos azúcares pero con pocos de los otros nutrientes, y convertir estos diversos azúcares a etanol, un compuesto antimicrobiano para el que esta levadura es muy tolerante. El vino se considera la primera bebida fermentada alcohólica, de hace unos 9000 años (Fay et al., 2019). Sin embargo, S. cerevisiae no es la única especie que interviene en la producción de bebidas alcohólicas, vinos incluidos, ya que también lo son otros Saccharomyces relacionados: S. uvarum, S. eubayanus, S. kudriavzevii y otros derivados híbridos (González et al., 2006).
Como el etanol es un buen preservativo natural frente a microbios no deseados, y adicionalmente tiene la capacidad de ser una sustancia adictiva, la fermentación de alimentos por levaduras se convirtió en una práctica bien extendida, y surgió independientemente en diversas civilizaciones en todo el mundo.
Para estudiar el probable origen de S. cerevisiae y la relación entre sus diversas cepas, en un reciente estudio de secuenciación y análisis de los SNP (polimorfismos de un solo nucleótido) de más de 600 cepas de esta especie (Duan et al., 2018) se ha visto que las cepas silvestres, de diversos continentes pero sobre todo de China, se separan casi todas de las domesticadas, excepto las aisladas en robles y otros Quercus, que son más cercanas a las de las cepas propias de vinos y de cervezas (Figura 1). Como vemos, las cepas domesticadas se distribuyen en dos ramas evolutivas. Una rama agrupa cepas de fermentaciones en estado sólido, sobre todo de Asia y en particular de China. Y la otra incluye las propias de fermentaciones líquidas como los vinos y cervezas y otros, además de las mencionadas de los Quercus. En base a todo ello, los autores (Duan et al., 2018) postulan un hipotético origen de la especie, y sobre todo de su domesticación, en China o en el este asiático en general.
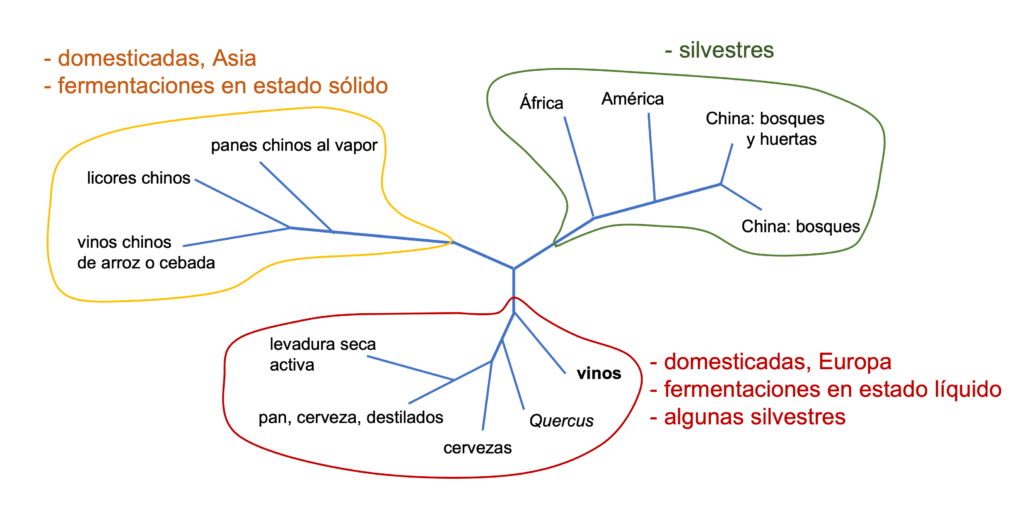
(adaptada de Steensels et al., 2019 y Duan et al., 2018)
Esta proximidad evolutiva de las cepas de S. cerevisiae del vino con las de los Quercus podría ser debida a que estos árboles fueran el origen silvestre de las cepas vínicas o también porque hubiera habido un retorno a la naturaleza de alguna de ellas (Steensels et al., 2019).
Las cepas de levaduras domesticadas de los diversos procesos en estado líquido, o industriales “europeas”, además de ser distintas a las silvestres se agrupan filogenéticamente con la aplicación industrial, ya sea cerveza o vino (Figura 2). Las cepas domesticadas de S. cerevisiae tienen reforzados unos rasgos que las hacen idóneas para el ambiente fermentativo, como la adaptación a concentración alta de azúcares, ambientes con poco O2 y tolerancia al etanol, pero también algunos rasgos específicos del producto. Por ejemplo, las levaduras cerveceras pueden metabolizar maltotriosa, azúcar específico de la malta, mientras que las levaduras vínicas tienen una buena resistencia a los agentes antimicrobianos empleados, el sulfato de cobre en el viñedo y el sulfuroso en la bodega.
Como vemos (Figura 2), las S. cerevisiae del vino tienen una domesticación más limitada que las cerveceras. Por ejemplo, estas últimas han perdido la capacidad de supervivencia fuera del entorno fermentativo, mientras que las vínicas pueden esporular y sobreviven en el entorno externo. Esto seguramente se debe a que la fermentación del vino solo ocurre una vez cada año, después de la vendimia, y el resto del año las levaduras deben sobrevivir a la bodega o al entorno en un ambiente más natural (Steensels et al., 2019).
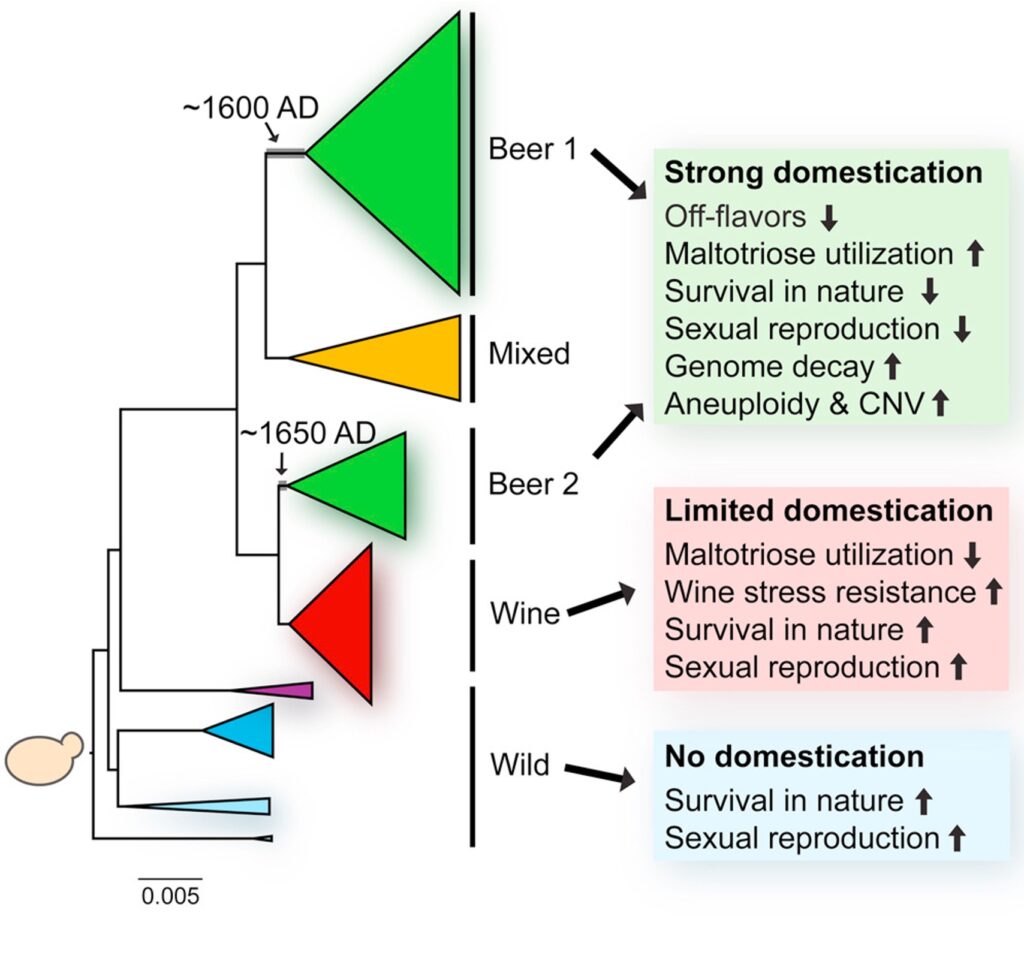
A estas diferencias entre las levaduras vínicas y cerveceras hay que añadir la mucho mayor diversidad génica de las cerveceras. De hecho, muchas de estas son S. cerevisiae que incluyen las llamadas de alta fermentación como en las cervezas ale y stout y muchas más de todo el mundo, pero justamente las cervezas de baja fermentación lager, que son el 94% del mercado mundial, son llevadas a cabo por S. pastorianus (o S. carlsbergensis, que es sinónimo), y por lo tanto otra especie. Este S. pastorianus es un híbrido alotetraploide de S. cerevisiae con la levadura criotolerante S. eubayanus, y se originó domésticamente hacia el siglo XV en Baviera sometiendo las cervezas a bajas temperaturas de cuevas de los Alpes. Por ello esta levadura actúa a 7-13ºC y en el fondo del fermentador (Libkind et al., 2011).
Todas las demás cervezas de alta fermentación son elaboradas con muy diversas S. cerevisiae, constituyendo un grupo polifilético con muchas ramas evolutivas, algunas de las cuales son filogenéticamente cercanas a las del sake, del vino y del pan. La diversidad de nucleótidos de las S. cerevisiae cerveceras es más del doble que la de las vínicas. Esta mayor diversidad seguramente se debe a la comentada utilización estacional de las levaduras vínicas a diferencia de las cerveceras todo el año, así como a los más intensos y diferentes regímenes de selección asociados a su elaboración (Gonçalves et al., 2016).
Con todo ello, tenemos la aparente paradoja de que las cervezas, como la mayoría de las consumidas son lager, y muchas de ellas producidas a gran escala industrial, parecen ser más estandarizadas comercialmente y por tanto más uniformes, y en cambio los vinos parecen ser más diversos, en contra de la mayor diversidad de cepas de levaduras cerveceras que vínicas.
Cambios genéticos hacia la domesticación de Saccharomyces cerevisiae en el vino
Al centrarnos en el vino, hay que recordar que al empezar la fermentación espontánea del mosto de uva las cepas de S. cerevisiae no son predominantes y existe un amplio espectro de otras levaduras, conocidas como no-Saccharomyces: Hanseniaspora, Pichia, Lachancea, Metschnikowia, Torulaspora y otros géneros (Fleet et al., 1984). Por efecto de la producción de etanol, S. cerevisiae acaba predominando y desplazando en número a las no-Saccharomyces. Aunque cada vez más se están viendo los beneficios de estas otras levaduras a nivel de aromas y se están utilizando como cultivos iniciadores adicionales a S. cerevisiae (Jolly et al., 2014), no se puede hablar de la domesticación de estos no-Saccharomyces porque no tienen hasta ahora un papel primordial en la vinificación y no se puede elaborar ningún vino exclusivamente con estas otras levaduras. Por tanto aquí nos limitaremos a S. cerevisiae.
Muchos de los mecanismos de cambios genéticos que han llevado a la domesticación de S. cerevisiae en la vinificación están presentes también en otras especies domesticadas, tanto microbianas como animales y plantas. El mecanismo más fácil de establecer como cambio genético son los SNP (polimorfismos de único nucleótido), debidas a mutaciones puntuales de un solo nucleótido, pero se ha visto que estos SNP representan tan solo una pequeña fracción de los relacionados con la domesticación de S. cerevisiae, y en cambio, la mayoría son arreglos estructurales del genoma, como los siguientes.
Las variaciones en el número de copias (CNV en inglés) son mutaciones muy frecuentes en muy diversos organismos, donde fragmentos de ADN de >1kb son repetidos, tienen varias copias, con número variable. El caso más conocido en las levaduras vínicas es el gen CUP1, que codifica para una metalotioneína, una proteína captadora de cobre. Algunas cepas pueden tener hasta 18 copias de este gen, con la que estas S. cerevisiae vínicas producen más cantidad de dicha proteína y por tanto son más tolerantes al cobre de la “mezcla de Burdeos”, usada como fungicida en los viñedos (Steensels et al., 2019).
Las reordenaciones cromosómicas con deleciones, inserciones o translocaciones de grandes fragmentos de cromosomas, a menudo causadas por transposones, son relativamente frecuentes en la evolución de las levaduras vínicas y de Saccharomyces en general. De hecho, estas reordenaciones que pueden afectar a un número importante de genes son una causa importante de especiación. Un ejemplo es la tolerancia al sulfito —el antibacteriano más usado en vinificación— debido al alelo SSU1-R, que es el producto sobreexpresado de una translocación entre los cromosomas 8 y 16, solo presente en las levaduras vínicas (Pérez-Ortín et al., 2002).
La hibridación interespecífica es otro mecanismo de cambios que ya hemos visto como origen de las levaduras domesticadas en las cervezas lager. Las especies de Saccharomyces pueden hibridar muy fácilmente entre ellas ya que estos híbridos pueden propagarse por mitosis tan eficientemente como sus progenitores, aunque raramente pueden realizar la meiosis y por lo tanto, casi nunca producen esporas viables (Sipiczki, 2008). Sin embargo, algunos de los linajes híbridos pueden acabar siendo reconocidos como especies. Los casos más conocidos son el mencionado S. pastorianus (sinónimo S. carlsbergenesis [S. cerevisiae x S. eubayanus]) de las cervezas a lager, y los S. bayanus (S. cerevisiae x S. eubayanus x S. uvarum) del vino (Figura 3). Sin embargo, S. bayanus es controvertido por sus similitudes con S. cerevisiae y se genera confusión en cuanto a los nombres de especies para alguna de las cepas comerciales más utilizadas (Sipiczki, 2008).
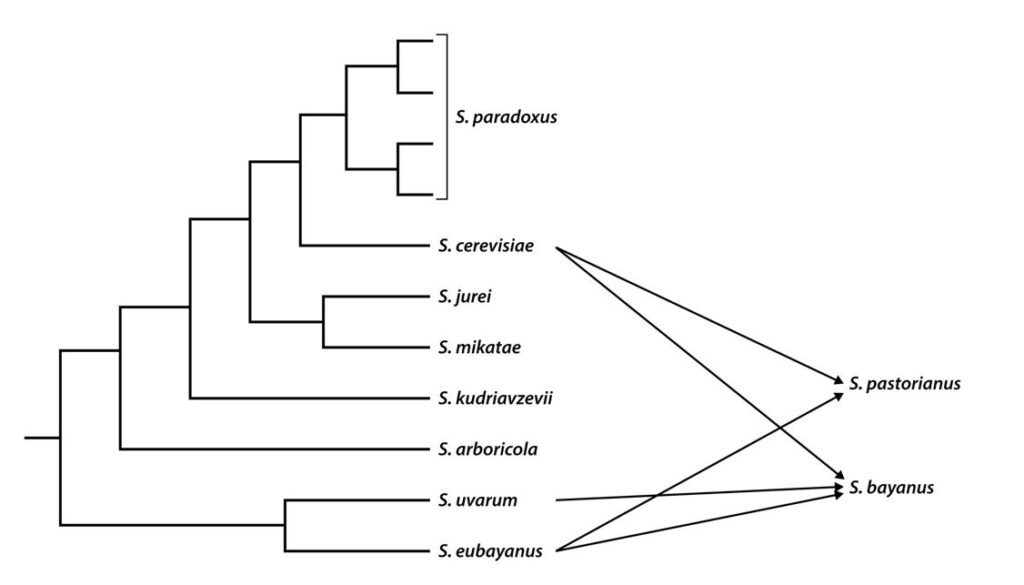
(modificada de Ono et al., 2020)
Los híbridos también han sido clave en la aparición de bastantes levaduras vínicas, sobre todo en relación con la posibilidad de fermentar a bajas temperaturas. Por ejemplo, algunas de las cepas comerciales de cultivos iniciadores que fermentan vino a 15ºC son híbridos “naturales” de S. cerevisiae con S. kudriavzevii con poliploidía parcial, aislados en 1979, que tienen la buena producción de etanol de S. cerevisiae y la tolerancia al frío de S. kudriavzevii (Erny et al., 2012).
De hecho, recientemente se está realizando en laboratorio la hibridación interespecífica de varios Saccharomyces vínicos —por ejemplo S. cerevisiae x S. uvarum—para conseguir cepas con mejores características relacionadas con caracteres poligénicos. La hibridación como método tiene la ventaja de no ser considerada una técnica GMO (organismo modificado genéticamente, en inglés) y por lo tanto las cepas obtenidas pueden ser transferidas rápidamente a la industria (García-Ríos et al., 2019).
La transferencia genética horizontal (TGH) es otro mecanismo muy importante evolutivamente en muchos organismos y en las levaduras vínicas también se conocen algunos casos que han contribuido a su domesticación. Un caso de éstos es el gen Fsy1 codificante del transportador de fructosa, que permite a S. cerevisiae la utilización de la fructosa al final de fermentación, después de haber consumido la glucosa. Es muy probable que este gen le fue transferido del similar de S. pastorianus, para el que presenta mucha homología (Galeote et al., 2010). Otro caso demostrado de TGH es el del gen Fot originario de otra levadura, Torulaspora microellipsoides, que le permite a S. cerevisiae una mejor incorporación de oligopéptidos, que le dan ventaja competitiva cuando las fuentes de nitrógeno son escasas (Marsit et al., 2015).
¿Levaduras del vino domesticadas o volvemos a las silvestres ?
A pesar de las ventajas en la fermentación del vino que ha supuesto el uso de estas levaduras evolucionadas, esto ha comportado una cierta uniformización en las características del vino, sobre todo a nivel de aromas, porque la mayoría de las levaduras utilizadas pertenecen a un solo linaje genético de S. cerevisiae domesticada. Como contraposición a esto y también por el deseo general de productos más “naturales” o ecológicos, últimamente hay una tendencia a utilizar microorganismos silvestres y/o autóctonos en lugar de las cepas “domesticadas”, o dejar que sean fermentaciones espontáneas. Esto ocurre con el vino pero también con la cerveza y otras bebidas y alimentos fermentados (Steensels et al., 2014). En el vino, la principal tendencia es, sobre todo, el uso de levaduras no-Saccharomyces, presentes de forma natural al inicio de la fermentación, como he comentado anteriormente, que dan perfiles aromáticos diferentes y muy interesantes (Padilla et al., 2016). Además, los avances en la comprensión de la domesticación combinados con las nuevas técnicas disponibles para la evolución dirigida de microorganismos posibilitarán la creación de nuevos linajes domesticados (Steensels et al., 2019).
Domesticación de las bacterias lácticas
El vino, junto con la cerveza y el pan, son los principales productos ligados a la domesticación comentada de la levadura S. cerevisiae, básicamente, con algún otro muy relacionado como hemos visto. Por lo tanto, tenemos unas pocas especies de un solo género con una gama limitada de productos. En cambio, las bacterias lácticas (BL) incluyen muchas especies y muy diversas que intervienen en muy diversos productos, tanto derivados de la leche como de vegetales y de otros alimentos (Tabla 1), muchos más que los tres básicos de las levaduras. Paradójicamente, y sin embargo, en el vino prácticamente solo tenemos una sola especie de bacteria láctica domesticada, Oenococcus oeni.
Las BL son bacterias gram-positivas de bajo G+C en su DNA, no esporuladas, de metabolismo fermentativo sin respiración, que producen ácido láctico como producto principal de la fermentación de carbohidratos. Desde el punto de vista taxonómico son el orden Lactobacillales, de la clase Bacilli, dentro del fílum Firmicutes. Sus hábitats naturales son muchas plantas, especialmente los frutos ricos en azúcares, y los animales, especialmente los mamíferos y en torno a sus leches, pero también en el interior de sus cuerpos, como en la microbiota de boca y digestivo.
Las BL son el mayor grupo de microorganismos en la producción de alimentos y bebidas fermentados a los que contribuyen en aroma, textura, seguridad y calidad. Su domesticación fue clave en la conservación de alimentos, dando lugar a muchos cultivos iniciadores para la producción de alimentos industriales fermentados.
A nivel industrial, los productos lácticos más conocidos son los lácteos o derivados de la leche (Tabla 1), donde las BL predominantes son Lactococcus y diversos géneros actuales derivados de la reclasificación del género Lactobacillus (Zheng et al., 2020). En estos dos grupos, la domesticación fue asociada a la pérdida de genes de rutas biosintéticas —de aminoácidos por ejemplo— y de estrés oxidativo, así como adquisición por TGH de genes para un crecimiento óptimo en el ambiente lácteo, como los de utilización de caseína y de lactosa (Bonham et al., 2017).
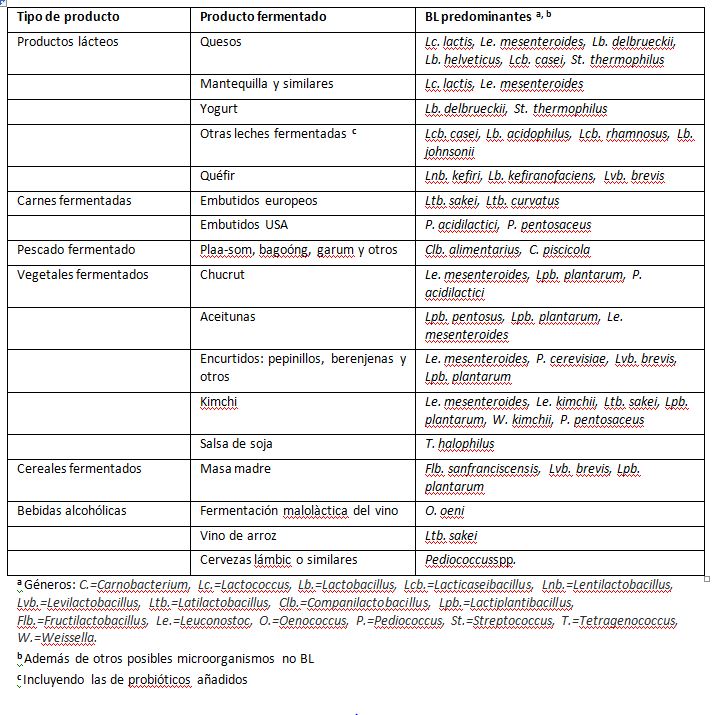
Tabla modificada y adaptada de Leroy & de Vuyst (2004). Se ha excluido Bifidobacterium, que estaba en el original, porque no es de Lactobacillales sino del fílum Actinobacteria. Se han actualizado los nuevos nombres del género Lactobacillus (Zheng et al., 2020)
Bacterias lácticas del vino, Oenococcus oeni y su domesticación
Al igual que con las levaduras, como las fermentaciones vínicas no son estériles, en el mosto puede haber muy diversas bacterias incluyendo las BL. De éstas, las más habituales durante la fermentación alcohólica, si bien en números bajos —inferiores a 104 UFC/mL— son Oenococcus oeni, Lactiplantibacillus plantarum, Lentilactobacillus hilgardii, Levilactobacillus brevis, Fructilactobacillus lindneri y Pediococcus parvulus (Capozzi et al., 2021).
Pero al final de la fermentación alcohólica (FA), cuando el contenido de etanol producido por las levaduras ya es superior a 10% (v/v) la gran mayoría de bacterias —incluyendo casi todas las BL— son inhibidas y prácticamente solo permanecen, y pueden crecer ligeramente, las cepas de O. oeni (Franquès et al., 2017) que llevan a cabo la fermentación maloláctica (FML) o conversión de L-málico a L-láctico. Esta FML tiene los beneficios de reducción de la acidez, mejora organoléptica y estabilidad microbiológica del vino (Bartowsky, 2008). Aparte de O. oeni, ocasionalmente en algunos vinos la FML puede ser llevada a cabo por otras BL, sobre todo por Lpb. plantarum (Krieger-Weber et al., 2020).
Por lo tanto, Oenococcus oeni es la bacteria del vino por excelencia, debido a su tolerancia al etanol y la capacidad de sobrevivir en un medio ácido y con polifenoles y con muy pocos nutrientes como es el vino. Su excepcionalidad está claro que va ligada a su domesticación —involuntaria para los humanos— y adaptación a este entorno artificial del vino durante estos últimos miles de años.
Cambios genéticos hacia la domesticación de Oenococcus oeni en el vino
Las bacterias gram-positivas con fermentación heteroláctica de azúcares y forma de coco que realizan la FML del vino fueron clasificadas como Leuconostoc oenos por Garvie (1967) por la semejanza con otros Leuconostoc. Sin embargo, las del vino se distinguen de los demás Le. por el crecimiento en medio ácido, la tolerancia al etano, y la pobre fermentación de carbohidratos, además de las estructuras diferenciadas de enzimas clave como la D-lactato deshidrogenasa o la glucosa-6-fosfato deshidrogenasa. El estudio filogenético del gen 16S rRNA por Yang & Woese (1989) reveló una línea bien diferenciada de estas bacterias del vino respecto a otras Leuconostoc y otras BL (Figura 4). Estos autores ya observaron que la secuencia del 16S rRNA de “Le. oenos“, además de ser muy distante del resto de Leuconostoc, tenía inusualmente alterada la composición en posiciones altamente conservadas. Por todo eso Dicks et al. (1995) propusieron el nuevo nombre de género y especie Oenococcus oeni.
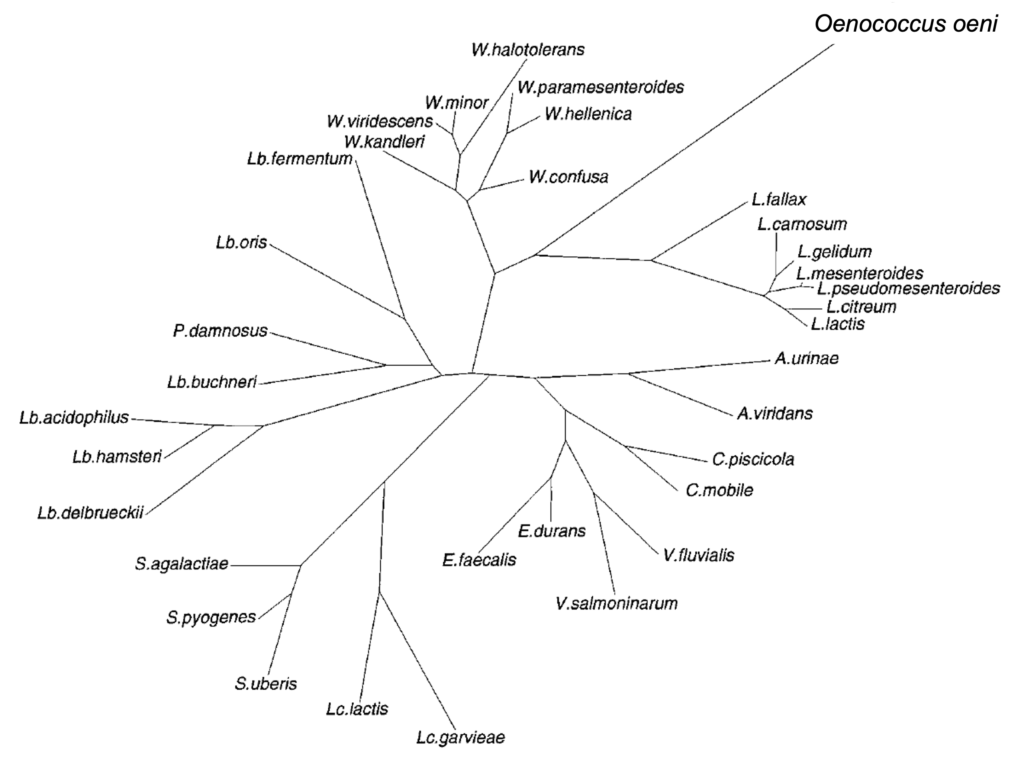
(adaptada de Dicks et al.,1995)
Esta rama filogenética muy larga y diferenciada de O. oeni es indicativa de la rápida evolución que esta bacteria ha experimentado en la domesticación y adaptación al vino. Las principales características genómicas de esta especie respecto a otras BL son de una reducción génica a todos los niveles (Makarova et al., 2006), lo que se corresponde con la menor capacidad biosintética (aminoácidos, vitaminas, etc.):
- El genoma de oeni es de 1,78 Mbp, el más pequeño de las BL, que van de 1,8 de varios Streptococcus a 3,3 de Lpb. plantarum.
- El número de operones rRNA de oeni es 2, el menor de todas las BL, ya que algunas como Lb. delbrueckiii tienen 9.
- El número de tRNA de oeni es 43, el menor de todas las BL, frente a 55-98 de las demás, relacionado con el menor crecimiento y la menor competitividad ecológica.
Además de la reducción génica, en Oenococcus se observan tasas más elevadas de mutaciones que en otras BL, es un género hipermutable (Tabla 2). La causa se debe a que están ausentes los genes mutS y mutL, que son genes de reparación de errores de apareamiento (MMR, mismatch repair), mientras que estos genes están presentes en todas las demás BL. El tipo de mutaciones más frecuente son las transiciones (GC→AT y AT→GC). Esto explica el alto nivel de polimorfismo alélico que probablemente condujo a su adaptación a los ambientes ácidos y con etanol (Marcobal et al., 2008). La alta mutabilidad debió de generar algunas mutaciones beneficiosas para ambientes muy restrictivos como es el vino, a diferencia de otras BL, como por ej. Leuconostoc o Pediococcus, que se encuentran en muy diversos hábitats. Además, el género Oenococcus es un ejemplo de cómo la carencia de los genes MMR puede llevar a la especiación.
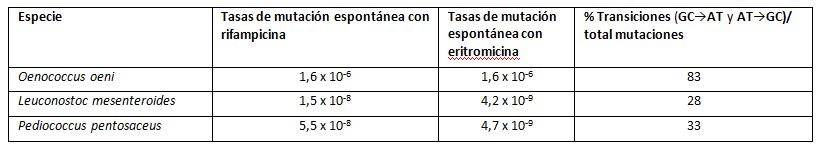
(adaptada de Marcobal et al., 2008)
Esta hipermutabilidad ha originado la gran diversidad de cepas de O. oeni que encontramos en todas las regiones vinícolas mundiales, y cuando se estudian sus genomas por diversos métodos moleculares como los SNP (polimorfismos de único nucleótido), se observan (Figura 5) agrupaciones de cepas relacionadas con el tipo de producto, como las de champán o de la sidra —donde también está presente esta bacteria—, resultado de la domesticación-adaptación a los diversos ambientes donde realiza la FML. También se encuentra O. oeni como BL predominante en la kombucha, la bebida de té fermentado por una comunidad de levaduras, bacterias acéticas y cepas específicas de O. oeni y otras BL (Lorentzen& Lucas, 2019).
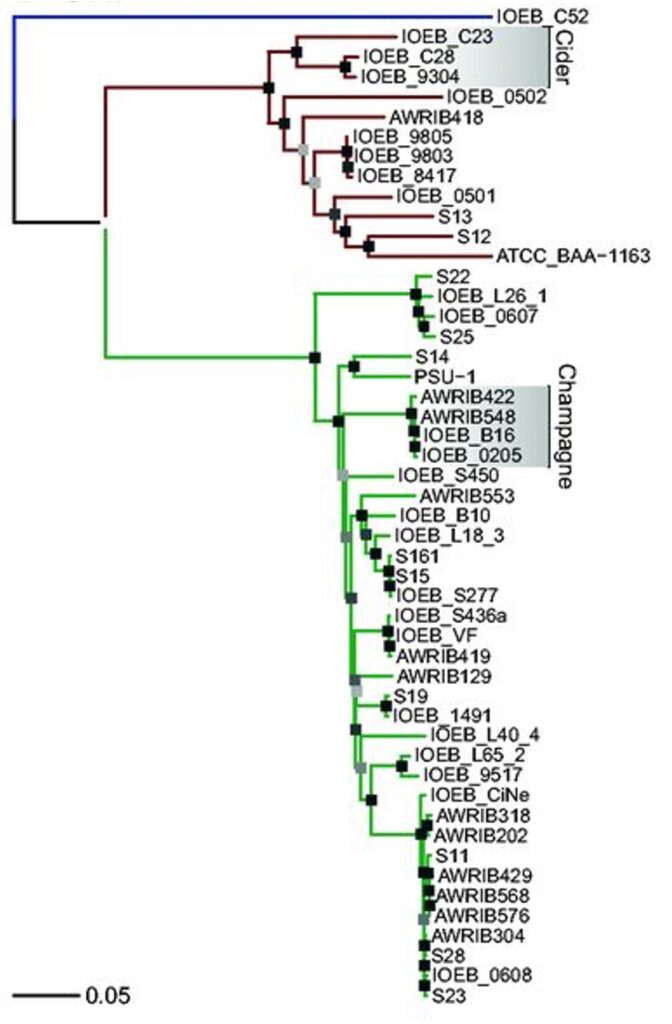
(adaptada de Campbell-Sils et al., 2015)
Oenococcus oeni, la bacteria domesticada presente casi solo en el vino
Esta es quizás la evidencia más clara de que O. oeni es una bacteria producto de la domesticación. De forma similar al caso de las levaduras, y en este caso se puede decir que aún con mayor certeza, es muy difícil encontrar O. oeni fuera del vino en ambientes naturales. Las poblaciones de BL en el mosto de uva son como máximo 104/mL, y O. oeni solo es una pequeña parte de estas. Hay muy pocos estudios en los que se les haya podido aislar de las uvas o del mosto. Destaca el reciente trabajo de Franquès et al. (2017) donde se consiguieron aislar varias cepas en el Priorat. Algunas de ellas fueron también aisladas en los mismos vinos y se confirmó la presencia del DNA de O. oeni en las mismas uvas.
A pesar de la escasa presencia de esta bacteria en las uvas y en el mosto, una vez la fermentación alcohólica está terminando y el vino contiene una cantidad significativa de etanol producido por las levaduras, los pocos O. oeni del inicio pueden haber alcanzado unas poblaciones suficientes para realizar la FML. Aparte del mosto, también es muy probable que las bacterias ya estén en la bodega, en las superficies o interior de los equipos, ya que se han detectado cepas comerciales de O. oeni en bodegas donde se había utilizado años antes (Lorentzen & Lucas, 2019).
Conclusión: la domesticación inconsciente de los microbios del vino generó dos muy excepcionales
La levadura Saccharomyces cerevisiae es la especie principal implicada en la fermentación alcohólica del vino y la bacteria láctica Oenococcus oeni lo es para la fermentación maloláctica. En cambio, antes de las fermentaciones respectivas apenas están ahí. Si no se inoculan cultivos iniciadores, los números de S. cerevisiae son muy bajos en el mosto, y O. oeni prácticamente no aparece en el vino hasta el final de la fermentación alcohólica. Por tanto, se puede decir que si los humanos no hubieran empezado a producir vino —y cerveza en el caso de las levaduras— hace algunos miles de años, estas dos especies seguramente no existirían. Son claramente microbios domesticados en el sentido que han aparecido evolutivamente en un tiempo relativamente breve como derivados de microbios previos que carecían de las características específicas de estos dos.
Como hemos visto, la excepcionalidad de Saccharomyces (“el hongo del azúcar”), y en particular S. cerevisiae, es sobre todo la capacidad de fermentar medios con muchos azúcares, convirtiéndolos en etanol en cantidades importantes, para el que esta levadura es muy tolerante. Esta producción de etanol es bioquímicamente excepcional y única entre los hongos y otros organismos. Véase al respecto el trabajo de Dashko et al. (2014) sobre la evolución de la fermentación alcohólica en las levaduras.
La excepcionalidad de O. oeni (“el coco del vino del vino”) también es evidente. Es una especie “nacida y moldeada” en el vino (Bech-Terkilsen et al., 2020), capaz de sobrevivir en un medio bien hostil, con etanol, pH bajo y muy pocos nutrientes. Subsiste gracias al L-málico y algunos restos de las células de levaduras, como es el caso de las manoproteínas (Balmaseda et al., 2021), y es resistente al estrés en estas condiciones extremas gracias a determinadas proteínas protectoras del choque del etanol, en los sistemas antioxidantes como el glutatión (Margalef-Català et al.. 2016) y también, en parte, debido a la acumulación de manganeso —también antioxidante y muy poco estudiado— que, además, es cofactor de la enzima maloláctica (Makarova et al., 2006). Por todo ello, O. oeni puede ser una especie modelo de estrés para las demás BL (Liu, 2002).
Bibliografía
Balmaseda A., Aniballi L., Rozès N., Bordons A., Reguant C.: (2021) Use of yeast mannoproteins by Oenococcus oeni during malolactic fermentation under different oenological conditions. Foods 10, 1540.
Bartowsky E.J.: (2008) Oenococcus oeni and malolactic fermentation —moving in to the molecular arena. Aust J Grape Wine Res 11: 174-187.
Bech-Terkilsen S., Westman J., Swiegers J., Siegumfeldt H.: (2020) Oenococcus oeni, a species born and moulded in wine: a critical review of the stress impacts of wine and the physiological responses. Aust J Grape Wine Res 26, 188-206.
Bonham K.S., Wolfe B.E., Dutton, R.J.: (2017) Extensive horizontal gene transfer in cheese-associated bacteria. Elife 6, e22144.
Campbell-Sills H., El Khoury M., Favier M., Romano A., Biasioli F. et al.: (2015) Phylogenomic Analysis of Oenococcus oeni Reveals Specific Domestication of Strains to Cider and Wines. Genome Biol Evol 7, 1506–1518.
Capozzi V., Tufariello M., De Simone N., Fragasso M., Grieco F.: (2021) Biodiversity of Oenological Lactic Acid Bacteria: Species- and Strain-Dependent Plus/Minus Effects on Wine Quality and Safety. Fermentation 7, 24.
Dashko S., Zhou N., Compagno C., Piškur J.: (2014) Why, when, and how did yeast evolve alcoholic fermentation?FEMS Yeast Res, 14, 826–832.
Dicks L.M.T., Dellaglio F., Collins M.D.: (1995) Proposal to reclassify Leuconostoc oenos as Oenococcus oeni [corrig.] gen. nov., comb. nov. Int. J. Syst. Bacteriol. 45, 395–397.
Duan S.F., Han P.J., Wang Q.M., Liu W.Q., Shi J.Y. et al : (2018). The origin and adaptive evolution of domesticated populations of yeast from Far East Asia. Nat. Commun. 9, 2690.
Erny C., Raoult P., Alais A., Butterlin G., Delobel P. et al.: (2012) Ecological Success of a Group of Saccharomyces cerevisiae/Saccharomyces kudriavzevii Hybrids in the Northern European Wine-Making Environment. Appl Environ Microbiol 78, 9.
Fay J.C., Liu P., Ong G.T., Dunham M.J., Cromie G.A. et al.: (2019) A polyploidy admixed origin of beer yeasts derived from European and Asian wine populations. PLoS Biol 17(3): e3000147.
Fleet G.H., Lafon-Lafourcade S., Ribereau-Gayon P.: (1984) Evolution of yeasts and lactic acid bacteria during fermentation and storage of Bordeaux wines. Appl Environ Microbiol 48, 1034–1038.
Franquès J., Araque I., Palahí E., Portillo M.C., Reguant C., Bordons A.: (2017) Presence of Oenococcus oeni and other lactic acid bacteria in grapes and wines from Priorat (Catalonia, Spain). LWT Food Sci Technol 81, 326-334.
Galeote V., Novo M., Salema-Oom M., Brion C., Valerio E. et al.: (2010) FSY1, a horizontally transferred gene in the Saccharomyces cerevisiae EC1118 wine yeasts train, encodes a high-affinity fructose/H+ symporter. Microbiology 156, 3754–3761.
Gallone B., Steensels J., Prahl T., Soriaga L., Saels V. et al: (2016) Domestication and divergence of Saccharomyces cerevisiae beer yeasts. Cell 166, 1397–1410.e1316.
García-Ríos E., Guillén A., De la Cerda R., Pérez-Través L., Querol A., Guillamón J.M.: (2019) Improving the Cryotolerance of Wine Yeast by Interspecific Hybridization in the Genus Saccharomyces. Front Microbiol 9, 3232.
Garvie E.I.: (1967) Leuconostoc oenos sp. nov. J Gen Microbiol 48, 431–438.
Goddard M.R., Greig D.: (2015) Saccharomyces cerevisiae: a nomadic yeast with no niche? FEMS Yeast Res. 15, 1–6.
Gonçalves M., Pontes A., Almeida P., Barbosa R., Serra M et al: (2016) Distinct Domestication Trajectories in Top-Fermenting Beer Yeasts and Wine Yeasts, Current Biology 26, 2750-2761.
González S.S., Barrio E., Gafner J., Querol A.: (2006) Natural hybrids from Saccharomyces cerevisiae, S. bayanus and S. kudriavzevii in wine fermentations. FEMS Yeast Res 6, 1221–1234.
Jolly N.P., Varela C., Pretorius I.S.: (2014) Not your ordinary yeast: non-Saccharomyce yeasts in wine production uncovered. FEMS Yeast Res, 14, 215–237.
Krieger-Weber S., Heras J.M., Suarez C.: (2020) Lactobacillus plantarum, a New Biological Tool to Control Malolactic Fermentation: A Review and an Outlook. Beverages 6, 23.
Leroy F., De Vuyst L.: (2004). Lactic acid bacteria as functional starter cultures for the food fermentation industry. Trends Food Sci Technol 15, 67–78.
Libkind D., Hittinger C.T., Valério E., Gonçalves C., Dover J. et al.:(2011) Microbe domestication and the identification of the wild genetic stock of lager-brewing yeast. Proceedings of the National Academy of Sciences, 108(35), 14539-14544.
Liu S.Q.: (2002) A review: Malolactic fermentation in wine — Beyond deacidification. J Appl Microbiol 92, 589–601.
Lorentzen M.P.G., Lucas P.M.: (2019) Distribution of Oenococcus oeni Populations in Natural Habitats. Appl Microbiol Biotechnol 103, 2937–2945.
Makarova K., Slesarev A., Wolf Y., Sorokin A., Mirkin B. et al.: (2006) Comparative genomics of the lactic acid bacteria. Proc Natl Acad Sci U S A. 103:15611–15616.
Marcobal A.M., Sela S.A., Wolf Y.I., Makarova K.S., Mills D.A.: (2008) Role of hypermutability in the evolution of genus Oenococcus. J Bacteriol 190, 564-570.
Margalef-Català M., Araque I., Weidmann S., Guzzo J. et al.: (2016) Protective roles of glutathione addition against wine-related stress in Oenococcus oeni. Food Res Internat 90, 8-15.
Marsit S., Mena A., Bigey F., Sauvage F., Couloux A. et al.: (2015) Evolutionary advantage conferred by an eukaryote-to-eukaryote gene transfer event in wine yeasts. Mol Biol Evol 32, 1695–1707.
Ono J., Greig D., Boynton P.J.: (2020) Defining and Disrupting Species Boundaries in Saccharomyces. Ann Rev Microbiol 74,477-495.
Padilla B., Gil J.V., Manzanares P.: (2016) Past and future of non-Saccharomyces yeasts: from spoilage microorganisms to biotechnological tools for improving wine aroma complexity. Front. Microbiol. 7, 411.
Pérez-Ortı́n J.E., Querol A., Puig S., Barrio E.: (2002) Molecular Characterization of a Chromosomal Rearrangement Involved in the Adaptive Evolution of Yeast Strains. Genome Res 12, 1533-1539.
Steensels J., Gallone B., Voordeckers K., Verstrepen K.J.: (2019) Domestication of industrial microbes. Curr Biol 29 R381 (Review).
Sipiczki M.: (2008) Interspecies hybridization and recombination in Saccharomyces wine yeasts. FEMS Yeast Res 8, 996-1007.
Steensels J., Snoek T., Meersman E., PiccaNicolino M. et al: (2014) Improving industrial yeast strains: exploiting natural and artificial diversity. FEMS Microbiol Rev 38, 947–995.
Yang D., Woese C.R.: (1989) Phylogenetic structure of the ‘‘Leuconostocs’’: an interesting case of a rapidly evolving organism. Syst Appl Microbiol 12, 145–149.
Zheng J., Wittouck S., Salvetti E. et al.: (2020) A taxonomic note of the genus Lactobacillus: Description of 23 novel genera, emended description of the genus Lactobacillus Beijerinck 1901, and union of Lactobacillaceae and Leuconostococaceae. Int J Syst Evol Microbiol 004107.






