Las contaminaciones microbiológicas del vino son muy perjudiciales por las considerables pérdidas económicas que representan para el sector vitivinícola y porque, si no son detectadas a tiempo, repercuten gravemente sobre la reputación y economía de la bodega productora. Esta situación es aún más grave en el caso de los vinos de crianza por el valor añadido de los mismos.
Brettanomyces bruxellensis (fig. 1) ha sido caracterizada históricamente como el principal agente responsable de la formación de fenoles volátiles como el 4-etilfenol, 4-etilguaiacol y tetrahidropiridinas, que producen aromas desagradables que alteran el vino sobre todo en las últimas etapas. Aunque no es necesariamente muy abundante, Brettanomyces se ha detectado en la uva y en el equipo de la bodega,1 y prolifera durante la crianza del vino y embotellado donde las poblaciones aumentan de manera lenta pero sin competencia, originando los principales riesgos.
Es destacable señalar que es muy probable que ninguna región vitivinícola esté libre de este microorganismo y que no hace falta un gran número de células de Brettanomyces para desarrollar la alteración, ya que por encima de 1000 células/mL la calidad organoléptica del vino está seriamente comprometida. Sin embargo, la relación presencia de Brettanomyces-deterioro del vino no es siempre evidente. En algunos estudios de nuestro grupo de investigación se han detectado especies alterantes como Brettanomyces tanto en vinos deteriorados como en vinos sin ninguna alteración aparente.2
Entre los motivos de su proliferación se encuentran:
- un estado sanitario deficitario de la uva de partida con lo que la concentración de levaduras iniciales sería inaceptablemente elevada;
- etapas prefermentativas, aunque se realicen a baja temperatura, ya que el crecimiento de B. bruxellensis solo se ve inhibido por debajo de 8ºC;
- exceso de nutrición durante la fermentación que produzca azucares residuales o aminoácidos y sales amoniacales que la levadura podrá usar para su crecimiento después de la fermentación;
- una relación inadecuada entre pH y contenido de sulfuroso, puesto que los pH elevados disminuyen la cantidad de SO2 molecular, bajando así su efecto antimicrobiano;
- una limpieza inadecuada de las barricas;
- presencia de oxígeno que estimule la producción de acidez volátil.
Detección en bodega
La detección en bodega de Brettanomyces se suele realizar mediante análisis sensoriales o de cromatografía gaseosa de los vinos en crianza para detectar la presencia de 4 etilfenol/ guayacol. Sin embargo, este método suele ser confirmativo, pues cuando se detecta el compuesto implica que la población de esta levadura es bastante elevada con lo que reparar la alteración resulta muy complicado.
Otros métodos de detección se basan en el empleo de medios de cultivo, como el DBDM,3 con agentes selectivos y diferenciales para evitar el crecimiento de otros microorganismos como levaduras, bacterias lácticas, etc., que poseen tasas de crecimiento mayor. Por otro lado, es necesario tratar volúmenes elevados de vino porque las poblaciones de Brettanomyces que suponen riesgo en vino se han determinado en 10 UFC/mL y el crecimiento lento de esta levadura hace que los tiempos de incubación sean de 8-10 días, posponiendo así la detección del problema.
Además, otro factor que complica su detección en el vino es la presencia de células en estado viable pero no cultivable (VPNC),4 efecto que viene dado por la adaptación de estos microorganismos a condiciones extremas del vino (concentraciones de etanol, polifenoles, SO2, ausencia de oxígeno, etc.). El fenotipo de VPNC se caracteriza por la incapacidad de las células de crecer en medio de cultivo aunque mantengan su actividad metabólica o celular de forma que pueden volver a ser cultivables cuando las condiciones ambientales sean favorables. Este fenómeno podría inducir a falsos negativos o infravaloración en placa de vinos con una considerable concentración de la levadura en este estado fisiológico, lo que supondría un riesgo para el vino analizado.2,4
Ventajas de las nuevas técnicas moleculares
Recientemente, la utilización de técnicas moleculares basadas en el material genético supone una ventaja respecto a las técnicas clásicas basadas en el cultivo. Las técnicas moleculares utilizadas incluyen análisis como la restricción del DNA mitocondrial,5 PCR-RFLP,6,7 PCR-RAPD,5 PCR con cebadores específicos8 y PCR anidada.9
Sin embargo, muchas de estas técnicas necesitan un paso de enriquecimiento previo para extraer el DNA de forma que son técnicas semicuantitativas. Por ello, se está tendiendo al uso de qPCR para la detección específica y cuantificación de B. bruxellensis directamente del vino. Aunque estas técnicas reducen el tiempo de análisis con respecto a las técnicas de cultivo y detectan incluso las células VPNC, al usar el DNA se detectan tanto células vivas como muertas, pudiendo dar lugar a una sobreestimación del número de levaduras en la muestra.2
Gracias a la introducción del bromuro monoazódico de etidio (EMA) o bromuro moazódico de propidio (PMB) combinados con la qPCR se han podido detectar únicamente las células vivas.2 Sin embargo, es necesario optimizar la concentración de EMA para ensayos diferentes y la concentración de etanol afecta los resultados.2
El RNA es considerado como un buen indicador de viabilidad ya que se degrada más rápidamente que el DNA10 aunque también es dependiente del gen usado para la detección. El problema principal de la qPCR a partir tanto de DNA como de RNA es que la qPCR solo detecta y cuantifica los microorganismos diana para los cuales hayamos diseñado cebadores de amplificación específicos y se desconoce si dichos microorganismos son los únicos implicados en el deterioro del vino.
En la actualidad, el estudio de la diversidad microbiana puede realizarse mediante una nueva técnica molecular denominada secuenciación masiva o HTS (de sus siglas en inglés high throughput sequencing). Consiste en la secuenciación de miles de secuencias por cada muestra tras la extracción directa de ácidos nucleicos de la matriz que se esté estudiando, en nuestro caso el vino. Existen diferentes tecnologías de secuenciación masiva11 cada una con sus ventajas y desventajas.12,13
El estudio de la composición microbiana se realiza de forma habitual mediante la amplificación de genes de interés taxonómico (normalmente el gen rRNA 16S para bacterias y el gen de rRNA 18S o el ITS para hongos) y puede ofrecer la proporción de los distintos grupos taxonómicos dentro de un alimento mediante la secuenciación de estos genes y su comparación con las bases de datos de referencia que proporcionarán la identificación de las distintas secuencias con lo que, además, tiene carácter cuantitativo.
En los últimos años, la secuenciación masiva se ha aplicado en prácticamente todos los campos de investigación de microbiología incluidos estudios sobre alimentos aunque el coste de estos análisis y el requerimiento de habilidades específicas bioinformáticas aún limitan su aplicación industrial.
Muchos de estos estudios tenían un marcado carácter de ecología microbiana (revisado en Ercolini).14 Por ejemplo, en el ámbito del vino se ha utilizado esta técnica para describir el microbioma del viñedo15-19 y las bodegas e instalaciones.15 Zarraonaindia et al.18 demuestran que la microbiota de la vid viene influenciada por las bacterias del suelo en el que se encuentran, las cuales son una importante fuente para las bacterias encontradas en la planta y uva. En esta misma línea, Portillo et al.19 demuestran que no solo la variedad de uva y el viñedo influyen en la microbiota residente en los racimos, sino que, además, factores como la orientación geográfica del viñedo marcan las diferencias en la composición bacteriana de las uvas dentro de la denominación de origen del Priorat (fig. 2).
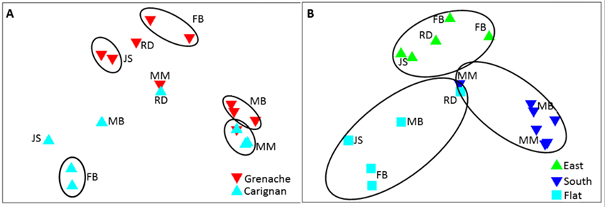
Brettanomyces no ha sido detectada entre las cientos de especies descritas como componentes del microbioma de la vid, mientras que Saccharomyces cerevisiae sí fue detectada, aunque poco
Por lo tanto, la identificación de los nichos naturales donde B. bruxellensis se encuentra es uno de los retos que limitan nuestro entendimiento sobre su biología y cómo se ha dispersado globalmente. Podríamos esperar que con el descenso en los costes de secuenciación próximamente se pueda revelar la presencia de esta especie fuera del ambiente de las bodegas.
| Hacia un mapa mundial de perfiles microbiológicos en regiones vitivinícolas
Un ejemplo de aplicación de HTS en el mundo comercial lo constituye Biome Makers, una empresa fundada por españoles con sede en San Francisco que ha apostado por introducir las técnicas genómicas en el mundo del vino. Actualmente ofrecen el estudio de suelo de viñedos que permite crear un mapa mundial de los perfiles microbiológicos en las diferentes regionales de vino y ver su evolución a lo largo de los años, con el objetivo de descifrar la influencia del suelo tanto en el proceso de vinificación como en el tipo de variedad de uva.
Esta empresa ha sido seleccionada por el primer programa de aceleración de empresas biotecnológicas en el área de la genética existente a escala mundial, promovido por la multinacional Ilumina (empresa líder en tecnología de secuenciación masiva). Con la filosofía según la cual, «el microbioma da una identidad única al terruño», WineSEQ propone identificar comunidades microbiológicas en diferentes regiones vitivinícolas del mundo, que permitan comparar entre distintas localizaciones para mejorar la calidad del vino. Más información en wineseq.com/ |
Las técnicas de HTS también se han aplicado ampliamente al estudio de la fermentación de alimentos o su deterioro microbiano14 y, recientemente, se ha puesto de manifiesto la capacidad y potencial de estas técnicas de HTS para poder detectar contaminaciones en alimentos y su posible trazabilidad en el entorno en el cual se procesan dichos alimentos.20 Hay que recordar que en muchos alimentos existen microorganismos pertenecientes al mismo género y en estos casos los estudios de HTS basados en secuencias muy cortas a nivel de género no serían de utilidad para diferenciar entre dichas especies. En estos casos, para obtener información a nivel de especie habría que tener como diana fragmentos más largos incluyendo más regiones variables de los genes taxonómicos o bien, complementar la técnica de HTS con alguna técnica de tipificación de especies como la RFLP (por ejemplo, Bokulich et al.21).
La ventaja indudable que las técnicas HTS aportarían al estudio del deterioro en el vino es que, al obtener miles de secuencias para una única muestra, se puede tener una descripción detallada de todos los microorganismos presentes en el deterioro y el cambio poblacional previo a la proliferación de un determinado microorganismo responsable de la alteración, con lo que podría tener carácter predictivo.
Además, las técnicas HTS pueden procesar cientos de muestras simultáneamente y se pueden realizar tanto a partir de DNA como de RNA, con lo que se podría tener información tanto de los microorganismos presentes, como los metabólicamente activos en el momento del deterioro, respectivamente. Por otro lado, mediante la metatranscriptómica (secuenciación masiva de todos los genes que se están transcribiendo en un determinado momento) se podría tener información de las interacciones metabólicas entre los distintos microorganismos implicados en el deterioro del vino. Todas estas técnicas basadas en secuenciación masiva, sin duda ofrecen una oportunidad para un estudio más detallado de los microbios responsables de la fermentación del vino y en su caso, de su posible deterioro.
Aunque las técnicas HTS no sean aplicables a corto plazo por las bodegas como rutina debido a los elevados costes de inversión en la tecnología necesaria y el grado de conocimiento específico que se necesita para realizar dicho análisis e interpretación de datos, sí que se pueden desarrollar servicios a bodegas, en la línea de la compañía Biome Makers ya citada, para que realicen análisis periódicos de poblaciones en vinos de crianza y así poder prevenir posibles contaminaciones microbianas.
Bibliografía
1. Renouf V., Strehaiano P., Lonvaud-Funel A.: Yeast and bacteria analysis of grape, wine and cellar equipments by PCR-DGGE. J Int Sci Vigne Vin 2007; 41: 51-61.
2. Andorrà I., Esteve-Zarzoso B., Guillamón J.M., Mas A.: Determination of viable wine yeast using DNA binding dyes and quantitative PCR. Int J Food Microbiol 2010; 144: 257-62.
3. Rodrigues N., Gonçalves G., Pereira-da-Silva S., Malfeito-Ferreira M., Lourerio V.: Development and use of a differential medium to detect yeast of the genera Dekkera/Brettanomyces. J Appl Microbiol 2001; 90: 588-99.
4. Millet V., Lonvaud-Funel A.: The viable but non-culturable state of microorganisms during storage. Lett Appl Microbiol 2000; 30: 136-41.
5. Martorell P., Barata A., Malfeito-Ferreira M., Fernández-Espinar M.T., Loureiro V., Querol A.: Molecular typing of the yeast species Dekkera bruxellensis and Pichia guilliermondii recovered from wine related sources. Int J Food Microbiol 2006; 106: 79-84.
6. Dias L., Pereira-da-Silva S., Tavares M., Malfeito-Ferreira M., Loureiro V.: Factors afecting the production of 4ethylfenol by the yeast Dekkera bruxelliensis in enological conditions. Food Microbiol 2003; 4: 377-84.
7. Esteve-Zarzoso B., Belloch C., Uruburu F. Querol A.: Identification of yeasts by RFLP analysis of the 5.8S rRNA gene and the two ribosomal internal transcribed spacers. Int J Syst Bacteriol 1999; 49: 329-37.
8. Campolongo S., Rantsiou K., Giordano M., Gerbi V., Cocolin L.: Prevalence and biodiversity of Brettanomyces bruxellensis in wine from Northwestern Italy. Am J Enol Vit 2010; 61 (4): 486-91.
9. Ibeas J.I., Lozano I., Perdigones F., Jiménez J.: Detection of Dekkera-Brettanomyces strains in sherry by a nested PCR method. Appl Environ Microbiol 1996; 62 (3): 998-1003.
10. Ivey M.L., Phister T.: Detection and identification of microorganisms in wine: a review of molecular techniques. J Ind Microbiol Biotech 2011; 38: 1619-34.
11. Shendure J., Ji H.: Next-generation DNA sequencing. Nat Biotechnol 2008; 26: 1135-45.
12. Suzuki S., Ono N., Furusawa C., Ying B.-W., Yomo T.: Comparison of sequence reads obtained from three next-generation sequencing platforms. PLoS One 2011; 6: e19534. doi:10.1371/journal.pone.0019534.
13. Liu L., Li Y., Li S., Hu N., He Y., Pong R., Lin D., Lu L., Law M.: Comparison of next-generation sequencing systems. J Biomed Biotechnol 2012: 251364.
14. Ercolini D.: High-throughput sequencing and metagenomics: moving forward in the culture-independent analysis of food microbial ecology. App Environ Microbiol 2013; 79 (10): 3148-55.
15. Bokulich N.A., Ohta M., Richardson P.M. Mills D.A.: Monitoring seasonal changes in winery-resident microbiota. PLoS One 2013; 8 (6): e66437.
16. Pinto C., Pinho D., Sousa S., Pinheiro M., Egas C., Gomes A.: Unravelling the diversity of grapevine microbiome. PLoS One 2014; 9: e85622.
17. Taylor M.W., Tsai P., Anfang N., Ross H.A., Goddard M.R.: Pyrosequencing reveals regional differences in fruit-associated fungal communities. Environ Microbiol 2014; 16: 2848-58.
18. Zarraonaindia I., Owens S.M., Weisenhorn P., West K., Hampton-Marcell J., Lax S., Bokulich N., Mills D.A., Martin G., Taghavi S., Van der Lelie D., Gilbert J.A.: The soil microbiome influences grapevine-associated microbiota. MBio 2015; 6: e02527-14.
19. Portillo M.C., Franquès J., Areque I., Reguant C., Bordons A.: Bacterial Diversity of Grenache and Carignan Grapes Surface from Different Vineyards at Priorat Wine Region (Catalonia, Spain). Int J Food Microb 2016; 219: 56-63.
20. De Filippis F., La Storia A., Villani F., Ercolini D.: Exploring the sources of beefsteaks contamination by culture-independent high-throughput sequencing. PLoS ONE 2013; 8: e70222.
21. Bokulich N. A., Joseph C. M. L., Allen G., Benson A. K., Mills D. A.: Next-Generation Sequencing Reveals Significant Bacterial Diversity of Botrytized Wine. PLoS ONE 2012; 7: e36357. doi:10.1371/journal.pone.0036357.
Bibliografía general
. Chattonnet P., Dubordieu D. Boidron J.: 1995. The influence of Brettanomyces/Dekkera sp. Yeast and lactic acid bacteria on the ethylphenol content of red wines. Am J Enol Vitic 1995; 46: 463-8.
. Claesson M.J., Wang Q., O’Sullivan O., Greene-Diniz R., Cole J.R., Ross R.P., O’Toole P.W.: Comparison of two next-generation sequencing technologies for resolving highly complex microbiota composition using tandem variable 16S rRNA gene regions. Nucleic Acids Res 2010; 38: e200. doi:10 .1093/nar/gkq873.
. Fuller C.W., Middendorf L.R., Benner S.A., Church G.M., Harris T., Huang X., Jovanovich S.B., Nelson J.R., Schloss J.A., Schwartz D.C., Vezenov D.V.: The challenges of sequencing by synthesis. Nat Biotechnol 2009; 27: 1013-23.
. Glenn T.C.: Field guide to next-generation DNA sequencers. Mol Ecol Resour 2011; 11: 759-69.
. Ribéreau-Gayon P., Dubourdieu D., Donèche B., Lonvaud-Funel A.: Handbook of Enology. The Microbiology of Wine and Vinifications. Vol 1. 2nd ed. West Sussex, England: John Wiley & Sons Ltd., 2016.