En muchos vinos después de la fermentación alcohólica efectuada por las levaduras tiene lugar la fermentación maloláctica, llevada a cabo por las bacterias lácticas. Éstas descarboxilan el ácido málico, dando ácido láctico y CO2, con lo cual disminuye la acidez del vino, pero además con la fermentación maloláctica también mejoran las características organolépticas y el vino se estabiliza microbiológicamente (Davis et al., 1988). Las cepas de bacterias lácticas aisladas de los vinos pertenecen a los géneros Lactobacillus, Leuconostoc, Pediococcus y Oenococcus, aunque en la mayoría de vinos la fermentación maloláctica es llevada a cabo por Oenococcus oeni (Van Vuuren & Dicks, 1993), especie antes conocida como Leuconostoc oenos (Dicks et al., 1995). Diversos estudios han demostrado que la resistencia de las bacterias lácticas a las condiciones del vino y su influencia sobre las calidades organolépticas dependen de la cepa bacteriana (Britz & Tracey, 1990). Por lo tanto, se precisan métodos que puedan diferenciar de manera fiable las diferentes cepas de O. oeni, para poder estudiar su dinámica de poblaciones en función de las diferentes condiciones del vino, tanto de las cepas autóctonas como de las inoculadas, y para comprobar si una cepa inoculada se impone y es ella la que está realizando la fermentación maloláctica.
Actualmente existen diversos métodos moleculares basados en el DNA, que permiten detectar e identificar si una bacteria láctica es de la especie Oenococcus oeni de forma precisa y fiable, a diferencia de los métodos fenotípicos tradicionales. Uno de estos métodos (PCR específica de especie) utiliza una PCR con cebadores (primers) específicos del gen del enzima maloláctico de O. oeni, que da un amplificado de DNA fácilmente detectable por electroforesis (Zapparoli et al., 1998). En nuestro laboratorio lo utilizamos para confirmar de forma rápida y precisa si los diversos aislados son O. oeni. Otra posibilidad es la detección directa de las bacterias de esta especie y de otras presentes en muestras de vino mediante la hibridación in situ con sondas específicas del 16S rRNA que están marcadas con fluorocromos, con la cual las bacterias pueden ser vistas y contadas en el microscopio de fluorescencia (Blasco et al., 2002).
Una vez se ha comprobado que un aislado es de la especie O. oeni, si se quiere tipificar la cepa es preciso recurrir a otros métodos moleculares. De estos, uno de los más eficaces es la macrorestricción del DNA total y separación de los fragmentos mediante electroforesis de campo pulsante (PFGE) (Daniel et al., 1993; Zapparoli et al. 2000). Aunque es una técnica muy reproducible y que da unos perfiles fáciles de analizar, es muy laboriosa. Por otro lado, otra técnica muy utilizada es la de los polimorfismos de DNA amplificado al azar (RAPD-PCR, de randomly amplified polymorphic DNA – polymerase chain reaction). Este método incorpora un único oligonucleótido cebador diseñado al azar que hibrida con muchos sitios del DNA y que al aplicar la PCR genera polimorfismos de los fragmentos de DNA (Williams et al., 1990). La RAPD-PCR es muy sensible, rápida, altamente discriminante y ha sido utilizada en diversas bacterias lácticas, incluyendo O. oeni (Zapparoli et al., 2000).
Sin embargo, este método a menudo tiene el inconveniente que su reproducibilidad no es muy buena, lo cual también lo hemos observado nosotros al hacer este estudio de cepas de vinificaciones, como se comenta a continuación. Por ello hemos desarrollado otro método (Multiplex RAPD-PCR) para diferenciar cepas de O. oeni, utilizando dos cebadores diferentes a la vez: un oligonucleótido de azar de 10 bases, junto con uno de los específicos del gen maloláctico, de 23 bases. El método ha dado muy buena reproducibilidad y ha sido aplicado con éxito para controlar las poblaciones de O. oeni durante tres vinificaciones en la misma bodega.
Aislamiento de bacterias de los vinos e identificación de la especie
Se tomaron muestras de vinificaciones en tinto de tres años sucesivos (1997, 1998 y 1999), realizadas en la bodega experimental de Mas dels Frares de la Facultad de Enología (Universitat Rovira i Virgili) en Constantí, Tarragona, en diversas condiciones (diferentes variedades, inoculación o no de levaduras, control de temperatura y otras variaciones). Con las muestras se inocularon placas de MRS suplementadas con 5 g/L de ácido d,l-málico, 10% de zumo de tomate, 0,5 g/L de l-cisteína, 50 mg/L de nistatina, 100 mg/L de azida sódica y 20 g/L de agar, a un pH 5,0. Después de incubar a 27°C, 30 colonias fueron escogidas al azar de cada muestra y crecidas aparte en MRS con 5 g/L de ácido d,l-málico y 6 g/L de fructosa. Los aislados fueron confirmados como bacterias lácticas si eran gram-positivas y catalasa-negativas.
De unos 1000 aislados de las tres vinificaciones, se procedió a centrifugar las células y extraer el DNA. Para ello se siguió el método de Persing et al. (1993) con algunas modificaciones, que consiste en primer lugar en resuspender las células en tampón Tris más EDTA, RNAsa y lisozima e incubarlo a 37°C durante una hora. A continuación, se consigue la lisis total de las células añadiendo una mezcla de tiocianato de guanidina, EDTA y sarcosil. Después se añade acetato amónico en frío y se agita sobre hielo unos 10 minutos. A continuación se realiza una extracción con cloroformo y 2-pentanol en proporción 24:1, y después de centrifugar, el DNA se precipita con isopropanol y finalmente se lava con etanol al 70%.
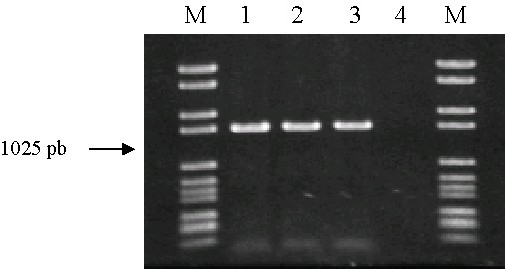
De estos DNA de todos los aislados, unos 800 fueron identificados como pertenecientes a la especie Oenococcus oeni mediante la mencionada técnica de PCR específica (Zapparoli et al., 1998), que amplifica un fragmento de 1025 parejas de bases del gen del enzima maloláctico de esta especie. Los cebadores son On1 (5′-TAATGTGGTTCTTGAGGAGAAAAT-3′) y On2 (5′-ATCATCGTCAAACAAGAGGCCTT-3′), y la amplificación de PCR es de 30 ciclos (45 segundos a 94°C, 2 minutos a 64°C y 2 minutos a 72°C). En la figura 1 se puede observar la banda correspondiente a este fragmento en la cepa tipo de O. oeni y en dos de las cepas aisladas, mientras que no aparece en la cepa tipo de Leuconostoc mesenteroides.
Desarrollo del método Multiplex RAPD-PCR para tipificar cepas de O. oeni
Al iniciar el estudio de tipificación de cepas de los casi 800 aislados identificados como O. oeni, empezamos aplicando un análisis de RAPD-PCR tal como se había realizado en un trabajo anterior con cepas de diferentes orígenes (Zaparolli et al. 2000). Se utilizaron por separado 4 cebadores cortos (de 10 a 15 bases, llamados M13, M14, 1283 y Coc), amplificando por PCR el DNA genómico total de cada aislado, y después efectuando electroforesis en geles de agarosa (1,4% en Tris-acetato con EDTA) y tiñéndolo con bromuro de etidio. Sin embargo, estos primeros resultados no fueron lo suficientemente discriminantes entre cepas para los tres cebadores M13, M14 y 1283, y la información que proporcionaban era redundante respecto al cebador Coc (5′-AGCAGCGTGG-3′), que era el más discriminante.
Al proceder a efectuar las RAPD-PCR solamente con el cebador Coc, se comprobó que pese a que distinguía muy bien las diferentes cepas, la reproducibilidad no era muy buena. Como se observa en la figura 2 (calles 1-4), los perfiles de bandas obtenidas para diferentes extracciones de DNA de la misma cepa son exactamente iguales si se hacen en la misma amplificación de PCR (calles 1 y 3, o 2 y 4), pero son ligeramente diferentes para diferentes amplificaciones (calles 1 y 2, o 3 y 4), con la cual no es suficientemente reproducible como para llevar a cabo un análisis exhaustivo de muchas cepas. De hecho, este es un problema habitual de la metodología RAPD.
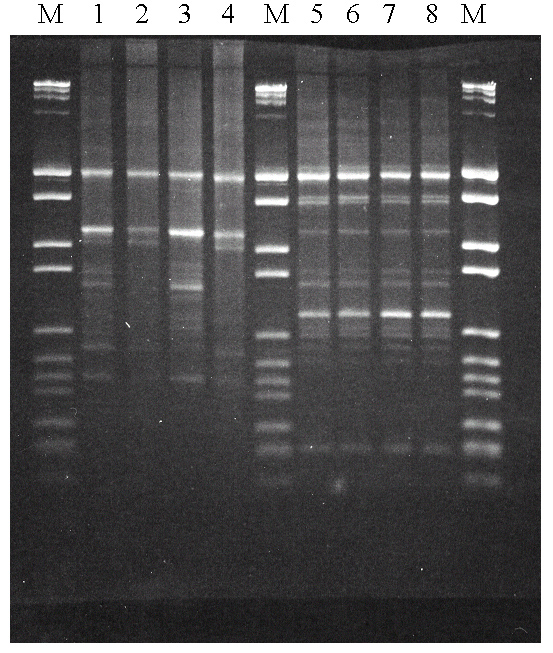
Para mejorar esta reproducibilidad, se procedió a ensayar el método Multiplex RAPD-PCR, consistente en efectuar la amplificación PCR con dos cebadores al mismo tiempo, método que ha funcionado con otras bacterias (Neilan, 1995, Holt & Cote, 1998). Realizamos ensayos combinando el cebador Coc con alguno de los otros mencionados y también con los utilizados para la PCR específica, On1 y On2. De todos ellos, la combinación de Coc con On2 fue la que dio mejores resultados, con una reproducibilidad excelente (figura 2, calles 5-8), y al mismo tiempo aparecían menos bandas de fragmentos de DNA amplificados, con la que es más fácil realizar el análisis y comparación de cepas.
Procedimos a optimizar este método Multiplex en condiciones de baja astringencia, probando diferentes temperaturas de hibridación y concentraciones de los cebadores, y los mejores resultados se obtuvieron con las condiciones siguientes. La mezcla de reacción PCR (20 µl) contenía 2 µl tampón PCR (10x), 0,25 mM de cada uno de los desoxinucleótidos (dATP, dCTP, dGTP y dTTP), 1 U DNA-polimerasa Taq, 3,5 mM MgCl2, 0,5 µM de Coc, 0,25 µM de On2 y 10 ng del DNA genómico. El perfil de amplificación con el termociclador fue 1 min a 94°C, 1 min a 40°C y 2 min a 72°C, repetido 30 ciclos, con una preincubación de 10 min a 94°C y una extensión de 10 min a 72°C. Los fragmentos de DNA fueron visualizados con bromuro de etidio mediante electroforesis en geles de agarosa.
Este método así optimizado ha resultado ser muy eficiente y el DNA de las cepas se puede analizar en tiempos relativamente cortos, de unas 6 horas. Por otro lado, es muy sensible, ya que permite distinguir cepas diferentes aunque sean muy parecidas.
Aplicación del Multiplex RAPD-PCR para diferenciar cepas de O. oeni de tres vinificaciones
En consecuencia, este método fue aplicado para tipificar los casi 800 aislados de O. oeni, que resultaron agrupados en 31 perfiles de bandas diferentes. Por lo tanto, éste sería el número total de cepas encontradas. Al inicio de las fermentaciones malolácticas se observaba un número variable de cepas, alrededor de 5, pero al acabar la fermentación maloláctica solamente había una cepa predominante, o dos en algunos casos. Aunque este número de cepas es relativamente bajo, cabe señalar que las diferentes vinificaciones de este estudio tuvieron lugar en la misma bodega, y por lo tanto los orígenes probables de inoculación espontánea de estos vinos con O. oeni serían los mismos.
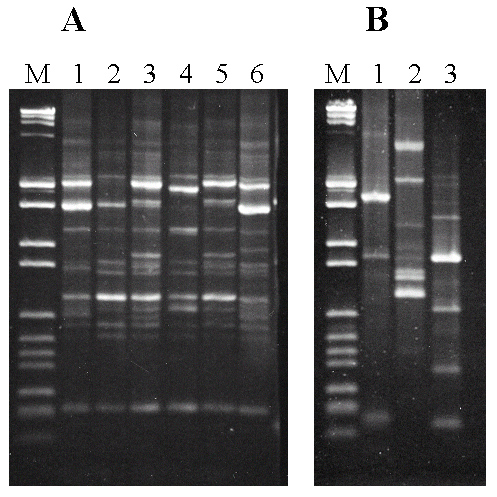
En la parte izquierda (A) de la figura 3 se muestran los perfiles de bandas de fragmentos de DNA amplificados con este método Multiplex RAPD-PCR, de las seis cepas predominantes en las diferentes vinificaciones. Como vemos, son perfiles relativamente parecidos, ya que hay algunas bandas (del mismo peso molecular) presentes en casi todas las cepas, pero la presencia o ausencia de muchas otras bandas (de un total de 50) permite distinguir con precisión las diversas cepas.
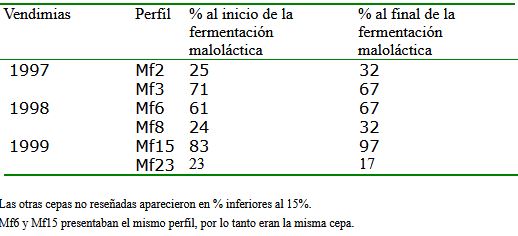
Por otro lado, en la tabla 1 vemos los porcentajes obtenidos para las 6 cepas mayoritarias mencionadas, al principio y al final de la fermentación maloláctica. La mayoría de cepas de O. oeni que se encontraron durante la fermentación alcohólica casi no se detectaban en la fermentación maloláctica, y viceversa, sugiriendo que unas cepas son sustituidas por otras a lo largo de las fermentaciones. Las cepas eran diferentes cada año, excepto Mf6 de 1998, cuyo perfil coincidía con el de Mf15 de 1999, que además fue la predominante estos dos años, y por lo tanto es la que ha realizado la fermentación maloláctica en la mayoría de los casos y en diferentes condiciones. Esta cepa debe ser la mejor adaptada a esta bodega y es una buena candidata para ser una estárter de la fermentación maloláctica.
.
Finalmente, también aplicamos este método Multiplex RAPD-PCR al DNA obtenido de tres especies diferentes de O. oeni, pero que también se pueden encontrar en el vino: Leuconostoc mesenteroides, Lactobacillus plantarum y Pediococcus pentosaceus. Como se puede observar en la parte derecha (B) de la figura 3, los perfiles de estas especies son completamente diferentes de los de las cepas de O. oeni, sin casi ninguna coincidencia de bandas. Esto confirma definitivamente la validez del método Multiplex que hemos desarrollado.
Conclusiones
En este estudio hemos diseñado y optimizado un nuevo método para diferenciar cepas de O. oeni, que incrementa notablemente la reproducibilidad y rapidez de los métodos convencionales de RAPD-PCR. Este método Multiplex consiste en utilizar dos cebadores a la vez en la PCR, uno de los cuales es específico de la especie O. oeni. El método ha sido validado controlando la dinámica de poblaciones de cepas a lo largo de la fermentación maloláctica de tres vinificaciones, y se ha visto que los casi 800 aislados de O. oeni eran 31 cepas diferentes. También se ha comprobado que al final de las fermentaciones malolácticas predominan una o dos cepas, y una de ellas ha sido la mayoritaria dos años, que por lo tanto debe ser la cepa que ha realizado la mayoría de las fermentaciones malolácticas eficazmente.
Por lo tanto este método seguramente será un buen instrumento para estudiar la dinámica de poblaciones de las bacterias lácticas durante la fermentación maloláctica, así como para hacer estudios de evaluación de cepas comerciales inoculadas, para comprobar su implantación y predominancia sobre las bacterias autóctonas, confirmando si el estárter inoculado es o no el que ha realizado la fermentación maloláctica.
Agradecimientos
Este trabajo ha sido realizado gracias a las ayudas ALI97-1077-C02-02 y AGL2000-0827-C02-02 del Ministerio de Ciencia y Tecnología. Cristina Reguant recibió una beca de la CIRIT y Ramon Carreté una beca del Ministerio de Educación y Cultura.
Bibliografía
. Blasco L., Pardo I., Ferrer S.: «Detection and identification of wine lactic acid bacteria by fluorescence in situ hybridization», Seventh FEMS Symposium on Lactic Acid Bacteria, Egmond an Zee, Països Baixos, 2002.
. Britz T.J., Tracey R.P.: «The combination effect of pH, SO2, ethanol and temperature on the growth of Leuconostoc oenos», J Applied Bacteriol 1990; 68, 23-31.
. Daniel P., de Waele E., Hallet J.N.: (1993) «Optimisation of transverse alternating field electrophoresis for strain identification of Leuconostoc oenos», Applied Microbiology and Biotechnology 1993; 38, 638-641.
. Davis C.R., Wibowo D., Fleet G.H., Lee T.H.: «Properties of wine lactic acid bacteria: their potential enological significance», American Journal of Enology and Viticulture 1998; 39: 137-142.
. Dicks L.M.T., Dellaglio F., Collins M.D.: «Proposal to reclassify Leuconostoc oenos as Oenococcus oeni [corrig.] gen. nov., comb. nov.», International Journal of Systematic Bacteriology 1995; 45: 395-397.
. Holt S.M., Cote G.L.: «Differentiation of dextran-producing Leuconostoc strains by a modified randomly amplified polymorphic DNA protocol», Applied and Environmental Microbiology 1988; 64: 3096-3098.
. Neilan B.A.: «Identification and phylogenetic analysis of toxigenic cyanobacteria by multiplex randomly amplified polymorphic DNA PCR», Applied and Environment Microbiol 1995; 61, 2286-2291.
. Persing D.H., Smith T.F., White T.J., Tenover F.C.: Diagnostic Molecular Biology, Principles and Application. Washington, D.C., ASM, 1993.
. Van Vuuren H.J.J., Dicks L.M.T.: «Leuconostoc oenos: a review», American Journal of Enology and Viticulture 1993; 44: 99-112.
. Williams J.G.K., Kubelik A.R., Livak K.J., Rafalski J.A., Tingey S.V.: «DNA polymorphisms amplified by arbitrary primers are useful as genetic markers», Nucleic Acids Research 1990; 18: 6531-6535.
. Zapparoli G., Reguant C., Bordons A., Torriani S., Dellaglio F.: «Genomic DNA fingerprinting of Oenococcus oeni strains by pulsed-field gel electrophoresis and randomly amplified polymorphic DNA-PCR», Current Microbiology 2000; 40: 351-355.
. Zapparoli G., Torriani S., Pesente P., Dellaglio F.: «Design and evaluation of malolactic enzyme gene targeted primers for rapid identification and detection of Oenococcus oeni in wine», Letters in Applied Microbiology 1998; 27: 243-246.