La fermentación vínica es un complejo ecosistema microbiano, en el que tanto levaduras como bacterias están tomando parte del proceso de transformación con sus actividades metabólicas. Los tradicionales métodos microbiológicos no permiten una comprensión completa de la ecología microbiana de estos sistemas complejos. Esto se debe principalmente a la capacidad de ciertos microorganismos de crecer en medios microbiológicos preferentemente con respecto a otros. Además, con estos métodos, las células con estrés o dañadas no pueden ser detectados en las placas. En los últimos 10 años se han desarrollado nuevos métodos basados en el análisis de los ácidos nucleicos (DNA y RNA) que se extraen directamente de la muestra sin necesidad de cultivo microbiano. El método que se utiliza a menudo en este tipo de estudios es la reacción en cadena de la polimerasa (PCR), junto con la desnaturalización de electroforesis en gel de gradiente (DGGE). Por otra parte, el desarrollo de herramientas moleculares para la caracterización de las cepas aisladas en la fermentación del vino permite una mejor comprensión de la dinámica de las cepas y la predominancia de unas u otras. El objetivo de este trabajo es mostrar las contribuciones más importantes al estudio de la ecología durante la fermentación vínica mediante el uso de herramientas moleculares, como la PCR-DGGE, para la caracterización de cepas.
Introducción
En las fermentaciones del vino conviven diferentes especies de levaduras, bacterias y hongos filamentosos. En cuanto a las levaduras, las principales responsables de la fermentación alcohólica pertenecen al género Saccharomyces (en particular la especie S. cerevisiae), mientras que las bacterias del ácido láctico o bacterias lácticas (BL) son las responsables de la fermentación maloláctica (Fleet, 1993). Dado que sus actividades metabólicas impulsan la transformación del mosto de uva al vino, es esencial comprender el comportamiento de los microorganismos durante la fermentación con el fin de obtener productos finales con las características organolépticas deseadas. Más concretamente, tras el desarrollo de microorganismos indeseables, se obtienen vinos con alta acidez volátil, sabores desagradables y con turbidez, debido al desarrollo de bacterias productoras de exopolisacáridos (Comi et al., 2006).
La dinámica microbiana de levaduras y bacterias se estudia desde los años 1970 (Barnett et al., 1972). Mediante el uso de los métodos clásicos, se realizan recuentos de microorganismos y se determina su diversidad en medios sintéticos con agar, pero en algunos casos se requiere un período de incubación en medio líquido, para promover el crecimiento de microorganismos específicos o seleccionarlos, ya que su aislamiento sería imposible sin este paso. El gran inconveniente de los métodos microbiológicos clásicos dependientes de cultivo es la imposibilidad de llegar a tener una visión precisa de la biodiversidad de estos ecosistemas complejos. Al utilizar los métodos de enriquecimiento y crecimiento en medios microbiológicos, la microbiota originalmente presente en la muestra es sometida a importantes cambios debido a la capacidad de ciertas especies para dominar el entorno y superar a los otros componentes microbianos. Por esta razón la población numéricamente menos importante, o en condiciones de estrés, apenas es recuperada e identificada. Con los métodos dependientes de cultivo hay un alto riesgo de errores de identificación de la ecología de los complejos ecosistemas microbianos (Hugenholtz et al., 1998).
A finales de los años 1990, los enfoques moleculares abrieron nuevas fronteras en la comprensión de la ecología microbiana. Estos métodos, llamados habitualmente como independientes de cultivo, son capaces de detectar e identificar microorganismos directamente en el sistema sin cultivarlos ni aislarlos, ya que analizan su DNA o RNA. Su novedad se basa en la extracción de los ácidos nucleicos directamente de las matrices, los cuales posteriormente son analizados por métodos capaces de poner de relieve la diversidad microbiana. Estudiando el DNA, es posible definir cuántas y cuáles son las especies microbianas presentes en una muestra específica, mientras que el estudio del RNA permite entender cuál es la parte metabólicamente activa de la población. Éste es un aspecto muy relevante en el caso de los alimentos fermentados, como el vino, donde es necesario estudiar las especies que son responsables de la transformación. Uno de los métodos independientes de cultivo más populares y más a menudo utilizado por los investigadores es la electroforesis en gel con gradiente desnaturalizante (DGGE), junto con la reacción en cadena de la polimerasa (PCR). En la PCR-DGGE, los ácidos nucleicos son sometidos a amplificación con cebadores universales, que son capaces de amplificar, en teoría, todo el DNA o el RNA de bacterias y levaduras presentes en un ecosistema específico. Después de la PCR, el amplicón estará constituido por una mezcla de diferentes productos de amplificación, que son más heterogéneas si la diversidad biológica en la muestra es compleja. Las secuencias de nucleótidos amplificados se separarán posteriormente con una electroforesis en un gel de poliacrilamida con un gradiente químico de los desnaturalizantes (urea y formamida). Cuando las moléculas de DNA encuentran el gradiente desnaturalizante capaz de abrir parcialmente (desnaturalizar) la doble hélice, se produce un cambio en la movilidad electroforética de la molécula, que en un momento dado hará que se detenga por completo en el gel. Dado que la desnaturalización del DNA depende de su secuencia, las diferentes moléculas de DNA tendrán una movilidad electroforética diferente. Con este enfoque entonces es posible diferenciar los microorganismos presentes en el mismo ecosistema, siempre y cuando presenten regiones de amplificación con secuencias diferentes. Para las fermentaciones alcohólica y maloláctica, diversos estudios han puesto de relieve que para las levaduras el bucle D1-D2 del gen del RNAr 26S (Cocolin et al., 2000) (fig. 1) y la región V7-V8 del gen del RNAr 16S de bacterias (López et al., 2003) son óptimas para ser utilizadas en el estudio de la ecología microbiana durante la vinificación.
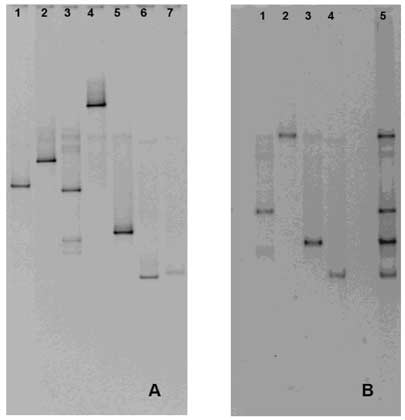
Figura 1B Perfiles DGGE obtenidos de cepas tipo de levaduras, y mosto al inicio de fermentación. Para la amplificación se utilizaron los cebadores NL1-NL2 (Cocolin et al., 2000) y las condiciones desnaturalizantes fueron de 30 a 60%. La electroforesis se hizo a 120V durante 4 h a 60ºC. Carriles: 1) Kluyveromyces thermotollerans DBVPG6480T; 2) Hanseniaspora uvarum DBVPG6717T; 3) Saccharomyces cerevisiae DBVPG6173T; 4) Brettanomyces bruxellensis DBVPG6707T; 5) muestra de mosto al inicio de fermentación
El desarrollo de métodos moleculares ha permitido también la diferenciación y caracterización de las cepas aisladas durante la fermentación. Ésta es una información importante en la que los investigadores están interesados porque puede dar una idea de la dinámica de cepas durante las fermentaciones. Mediante el uso de métodos moleculares de caracterización o tipificación es posible por ejemplo definir qué cepa es capaz de llevar a cabo la fermentación y en el caso de la adición de cultivos iniciadores, definir si la cepa inoculada será capaz de hacerse cargo del proceso y convertirse en la cepa dominante.
En este trabajo se presentan algunos ejemplos de métodos moleculares independientes de cultivo, así como de tipificación de los microorganismos aislados de la fermentación vínica.
PCR-DGGE en la fermentación vínica
La PCR-DGGE es uno de los métodos más frecuentemente utilizados para la definición de la ecología microbiana en la fermentación del vino. El primer estudio que utilizó PCR-DGGE en este sector fue publicado por Cocolin et al. (2000). En él se estudió la dinámica microbiana de levaduras enológicas, como Saccharomyces cerevisiae, Metschikowia pulcherrima, Candida ethanolica y Kloekera apiculata a lo largo de fermentaciones de laboratorio. Un aspecto importante resaltado en este estudio fue la posibilidad de controlar las poblaciones de otras especies de levadura que fueran al menos 0,01% con respecto a la Saccharomyces dominante, definiendo de esta manera el límite de sensibilidad del método. El protocolo se utilizó para estudiar la ecología de levaduras durante la fermentación comercial de un vino dulce, elaborado a partir de uvas deliberadamente infectadas con Botrytis cinerea. Se identificaron varias levaduras no-Saccharomyces, tales como Metschnikowia sp. y Pichia anomala (Cocolin et al., 2001b). Además, se observó a lo largo de la fermentación una traza constante de una especie de Candida. A raíz de los estudios de la fermentación del mencionado vino dulce, se observó que el perfil molecular de varias especies de levadura estaba presente en los geles de DGGE geles incluso cuando sus colonias no eran visibles en los medios de cultivo de agar (Mills et al., 2002). Este aspecto fue particularmente importante para una especie de Candida, posteriormente identificada como C. zemplinina (Sipiczki, 2003). Teniendo también en cuenta los resultados de dot blot de RNA obtenidos en este estudio, fue posible demostrar que las células metabólicamente activas, aunque no cultivables, podrían estar presentes durante la fermentación alcohólica. Después de estas primeras aplicaciones, la PCR-DGGE fue usada extensivamente para estudios de enología. Cocolin et al. (2002) lo utilizaron para controlar fermentaciones continuas, donde se destacó la presencia de no-Saccharomyces solamente en la etapa inicial de puesta a punto del fermentador, mientras que durante el resto del proceso, Saccharomyces fue el único microorganismo detectado en los geles. Renouf et al. (2006a) utilizaron el DGGE para seguir la ecología de tres bodegas francesas durante la fermentación alcohólica y la maloláctica. En los tres casos se pudo detectar la presencia constante de Brettanomyces bruxellensis, una de las levaduras enológicas más controvertidas, capaz de persistir en el vino acabado.
Mientras que la PCR-DGGE ha sido un método frecuentemente utilizado para estudiar la ecología de levaduras, sólo unos pocos estudios han sido publicados sobre la ecología bacteriana durante la elaboración del vino. Un problema importante que había que resolver era la no-especificidad de los cebadores utilizados para amplificar los DNA bacterianos. Como fue demostrado por López et al. (2003), varios cebadores específicos “bacterianos” también amplificaban DNA de levaduras, mohos y plantas. Un cuidadoso estudio de las secuencias de los genes ribosomales dio como resultado la definición de cebadores altamente específicos para bacterias, que podrían ser utilizados para el análisis de DGGE. Además, se han utilizado otras dianas de amplificación con el mismo propósito. La subunidad β de la RNA polimerasa (rpoB) se ha propuesto como gen alternativo al utilizar el DGGE (Dahllof et al., 2000). En concreto, Renouf et al. (2006b) utilizaron cebadores para la rpoB para estudiar la ecología bacteriana en diferentes bodegas. Este trabajo puso de relieve que se detectaron diferencias en la ecología bacteriana sólo al inicio de la fermentación maloláctica, mientras que al final solamente estaba presente Oenococcus oeni. Por último, en 2008, Urso et al., publicaron un artículo sobre la ecología de levaduras durante la fermentación de un vino dulce en Italia, con uvas secadas de forma natural en la planta. El comienzo de la fermentación fue dominado por hongos filamentosos, tanto a nivel de DNA como de RNA, pero después del estrujado, en el mosto, las levaduras, como Kloeckera y Candida, seguidas por S. cerevisiae, fueron capaces de dominar la fermentación.
Caracterización molecular de las cepas de levadura aisladas durante la vinificación
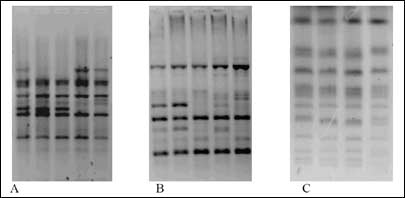
Un campo importante de investigación, dirigido a aclarar la compleja interacción de levaduras y bacterias durante la fermentación del vino, es la caracterización molecular de las especies aisladas durante el proceso de transformación. En los últimos 20 años se han propuesto varios métodos y la literatura está llena de trabajos que describen el uso de estos métodos para diferenciar cepas de la misma especie. La electroforesis de campo pulsante (PFGE), el análisis de restricción del DNA mitocondrial, el DNA polimórfico amplificado al azar (RAPD), la Sau-PCR (con el enzima de restricción Sau3A) y los minisatélites son los métodos de caracterización más utilizados en estudios de ecología durante la fermentación vínica. Estos métodos se combinaron en un estudio publicado por Cocolin et al. (2004), con el fin de investigar la diferencia genética entre los aislados de S. cerevisiae en bodegas modernas y antiguas. Mediante el uso de RAPD, PFGE y Sau-PCR, fue posible diferenciar las cepas estudiadas en función de su lugar de aislamiento (fig. 2). Por otra parte, con el complemento de análisis fisiológicos, se demostró que las cepas aisladas de las bodegas modernas habían modificado caracteres específicos bajo la presión del medio ambiente bodega (efecto bodega). La técnica de amplificación RAPD también ha sido recientemente utilizada por Romano et al. (2008) para investigar la biodiversidad de cepas silvestres de S. cerevisiae, y optimizar la calidad del vino (Figura 3). Además por otra parte, Urso et al. (2008) fueron capaces de seguir la dinámica de cepas de S. cerevisiae durante la fermentación de vinos dulces inoculados con cultivos iniciadores específicos. Se comprobó que en un caso, el cultivo iniciador no fue capaz de realizar la transformación, que estaba dominada por cepas silvestres procedentes de la uva utilizada.

Conclusiones
El desarrollo tecnológico y los avances científicos en el campo de la biología molecular han llevado a la creación de instrumentos de análisis cada vez más precisos y exactos para el estudio de sistemas microbianos complejos. La aplicación de este enfoque permite una mejor comprensión de los procesos microbianos, que previamente habían sido caracterizados por los métodos tradicionales de cultivo, pero no aclarados por completo. Mediante el uso de métodos independientes de cultivo y la caracterización molecular de las cepas aisladas, ha sido posible una mejor comprensión de esta compleja transformación. Es importante destacar que la posible presencia de células viables pero no cultivables, especialmente en el caso de la levadura contaminante Brettanomyces bruxellensis, debe ser cuidadosamente evaluada debido a la conexión directa de estos microorganismos y sus actividades metabólicas con la calidad organoléptica del producto final.
El uso de métodos moleculares en las fermentaciones vínicas ha permitido un conocimiento más profundo de las interacciones entre diferentes especies de levaduras y entre éstas y las bacterias, de manera que se pueden prever posibles intervenciones, con el fin de controlar el proceso y obtener un vino con óptimas características organolépticas y sensoriales.
Bibliografía
.Barnett J.A., Delaney M.A., Jones E., Magson A.B., Winch B. The numbers of yeasts associated with wine grapes of Bordeaux. Archives of Microbiology 1972; 83: 52-55.
.Cocolin L., Bisson L.F., Mills D.A. Direct profiling of the yeast dynamics in wine fermentations. FEMS Microbiology Letters 2000; 189: 81-7.
.Cocolin L., Heisey A., Mills D.A. Direct identification of the indigenous yeasts in commercial wine fermentations. American Journal of Viticulture and Enology 2001; 52: 49-53.
.Cocolin L., Manzano M., Rebecca S., Comi G. Monitoring of yeast population changes during continuous wine fermentation by molecular methods. American Journal of Viticulture and Enology 2002; 53: 24-27.
.Cocolin L., Pepe V., Comitini F., Comi G., Ciani M. Enological and genetic traits of Saccharomyces cerevisiae isolated from former and modern wineries. FEMS Yeast Research 2004 5: 237-45.
.Comi G., Manzano M., Cocolin L. Le alterazioni microbiche dei vini. En: M. Vincenzini, P. Romano, G.A. Farris (eds.) Microbiologia del vino. Casa Editrice Ambrosiana, Milán, I, 2006: pp. 315-346.
.Dahllof I., Baillie H., Kjelleberg S. rpoB-based microbial community analysis avoids limitations inherent in 16S rRNA gene intraspecies heterogeneity. Applied and Environmental Microbiology 2000; 66: 3376-80.
.Fleet, G. H. (1993) Wine microbiology and biotechnology. Ed. Harwood Academic Publishers GmbH, New York, USA.
.Hugenholtz P., Goebel B.M., Pace N.R. Impact of culture-independent studies on the emerging phylogenetic view of bacterial diversity. Journal of Bacteriology 1998; 180: 4765-4774.
.Lopez I., Ruiz-Larrea F., Cocolin L., Orr E., Phister T., Marshall M., VanderGheynst J., Mills D.A. Design and evaluation of PCR primers for analysis of bacterial populations in wine by denaturing gradient gel electrophoresis. Applied and Environmental Microbiology 2003; 69: 6801-6807.
.Mills D.A., Johannsen E., Cocolin L. Yeast diversity and persistence in botrytis-affected wine fermentations. Applied and Environmental Microbiology 2002; 68: 4884-4893.
.Renouf V., Falcou M., Miot-Sertier C., Perello M.C., De Revel G., Lonvaud-Funel A. Interactions between Brettanomyces bruxellensis and other yeast species during the initial stages of winemaking. Journal of Applied Microbiology 2006a; 100: 1208-1219.
.Renouf, V.; Classe, O.; Miot-Sertier, C.; Lonvaud-Funel, A. Lactic acid bacteria evolution during winemaking: use of rpoB gene as a target for PCR-DGGE analysis. Food Microbiology 2006b; 23: 136-45.
.Romano P., Capace A., Serafino V., Romaniello R., Poeta C. Biodiversity of wild strains of Saccharomyces cerevisiae as tool to complement and optimize wine quality. World Journal of Microbiology and Biotechnology 2008; 24: 1797-1802.
.Sipiczki, M. Candida zemplinina sp. nov., an osmotolerant and psychrotolerant yeast that ferments sweet botrytized wines. International Journal of Systematic and Evolutionary Microbiology 2003; 53: 2079-83.
.Urso R., Rantsiou K., Dolci P., Rolle L., Comi G., Cocolin L. Yeast biodiversity and dynamics during sweet wine production as determined by molecular methods. FEMS Yeast Research 2008; 8: 1053-1062.






