Las levaduras enológicas no deberían guardar sus secretos por mucho tiempo más. Los progresos realizados desde la llegada de la biología molecular en los años ochenta han llevado a un arsenal de herramientas y enfoques que permiten a los microbiólogos de hoy día abordar problemas considerados durante mucho tiempo como inaccesibles. A raíz de los conocimientos surgidos del análisis de los genomas, han visto la luz todo un conjunto de nuevas técnicas, en particular las conocidas como ómicas (para expresar el carácter global de la misma manera que el genoma se refiere a todos los genes de un organismo), que actualmente ya están disponibles.
Durante mucho tiempo los científicos se han tenido que limitar a la descripción del comportamiento de fermentación de las levaduras y a una comprensión a nivel macroscópico. Esto se ha logrado en diversos contextos por muchos grupos de investigación y ha permitido progresos considerables en el control de la fermentación alcohólica.
Sin embargo, el acceso a mecanismos más íntimos que subyacen en el comportamiento de los microorganismos ha sido difícil durante tiempo. Por ello los enólogos se han hecho siempre elementales preguntas tales como: ¿por qué esta levadura tiene más dificultades para acabar la fermentación que otra levadura determinada? ¿Por qué esa otra levadura da más aromas afrutados a baja temperatura? Estas preguntas sencillas no tienen respuesta la mayoría de veces. Es previsible que, en los próximos años, veamos notables avances especialmente en el conocimiento de los mecanismos genéticos, fisiológicos y bioquímicos que subyacen a estas propiedades. Por supuesto, cuando se dice conocimiento de los mecanismos, significa también entender los mecanismos de acción para intervenir y modular los efectos en la dirección deseada. Hay diversas maneras de mejorar las cepas, y se pueden considerar, según el caso, estrategias de genética clásica o de ingeniería genética para obtener una cepa con características nuevas.
El genoma: la información de base
En 1996 se dio un paso importante con la secuenciación del genoma completo por primera vez de la levadura Saccharomyces cerevisiae (Goffeau et al., 1996). Ello reveló la disposición de unos 6000 genes en sus 16 cromosomas. Afortunadamente para la enología, este microorganismo preferido también había llegado a ser un organismo modelo, al ser el eucariota más sencillo, un organismo en el que es fácil adquirir los conocimientos básicos, y en torno al cual se habían movilizado muchos equipos de investigación. Se consiguió una producción considerable de datos que llevaron a la adjudicación de una función para un 80% de los 6000 genes identificados en este microorganismo. Si bien la obtención de la secuencia fue un paso importante, después ha sido necesario un largo trabajo, aún sin terminar, con el fin de asignar una función a cada uno de los genes.
Durante más de 10 años la comunidad de microbiólogos se ha basado en los datos de un único genoma de levadura, de una única cepa de laboratorio. Para los que trabajan con levaduras enológicas, era evidente que este genoma no se correspondía exactamente con el de sus cepas industriales preferidas. En efecto, diversas observaciones indicaban divergencias entre el genoma de la cepa de laboratorio y el de las enológicas. Recientemente la evolución de las capacidades de secuenciación ha permitido desvelar las diferencias genómicas entre las cepas enológicas y sus homólogas de laboratorio o las de otras industrias. Mientras que para obtener la secuencia de la primera cepa se habían necesitado casi 10 años de esfuerzos, con las nuevas tecnologías de secuenciación se puede obtener un genoma completo de levadura en un solo día (!). En la actualidad han sido descifrados genomas de más de 100 cepas diferentes (Liti et al., 2009). Ello ha permitido precisar las relaciones entre las cepas de diversos orígenes (vino, pan, etc.), analizando la distribución de unas 30 000 a 50 000 mutaciones que permiten distinguir entre dos clones. También se ha constatado que nuestras cepas enológicas forman un grupo que se diferencia claramente de las cepas de otros ambientes.
El resultado más sorprendente ha sido revelado por un análisis detallado del genoma completo de la cepa enológica EC1118, muy utilizada en vinificación (Novo et al., 2009). De hecho, este trabajo ha puesto en evidencia la existencia de genes nuevos, ausentes en las cepas de laboratorio, procedentes de otras especies diferentes de Saccharomyces cerevisiae, sobre todo de Zygosaccharomyces. Estos nuevos genes se habrían transmitido en sucesos improbables, de una especie a otra, por mecanismos mal conocidos de transferencia «horizontal». Así pues, en su historia las levaduras enológicas tienen uno o varios ancestros que han adquirido algunos genes debido a la compañía de cepas de otras especies en las cubas. Al mismo tiempo, estos resultados ponen de relieve la fragilidad de la barrera entre especies en términos de transferencia de genes. Antes pensábamos que esta barrera era muy sólida, pero no parece ser el caso.
Enfoques de genómica funcional
Si durante mucho tiempo las preguntas fueron del orden de «¿cuál es el gen responsable de esta función?», una vez que se conoce la secuencia del genoma, se plantean las preguntas inversas, como la recurrente: ¿para qué sirve este gen? , o mejor aún, ¿este gen tiene un papel importante en la fermentación alcohólica del vino? Se han desarrollado diversas estrategias y metodologías para responder a estas preguntas. La más sencilla consiste en inactivar un gen para evaluar el efecto de su ausencia en el comportamiento de la levadura. Esto es fácilmente realizable en S. cerevisiae. Para ello basta con construir un fragmento de DNA que contenga un gen de resistencia a una droga, flanqueado por dos partes similares al gen a estudiar. Mediante transformación y recombinación homóloga el fragmento en cuestión va a reemplazar el gen inicial en algunas células, que serán fácilmente seleccionadas porque son resistentes a la droga. De hecho, tras un gran esfuerzo colectivo, se construyó una colección de 6000 mutantes que contenían cada uno de los 6000 genes de la levadura inactivada. Los clones obtenidos han sido ampliamente utilizados para acceder a las funciones de los genes y evaluar su importancia en las condiciones enológicas. Sin embargo, estas colecciones tienen el inconveniente de haber sido desarrolladas con cepas de laboratorio, que no poseen todas las propiedades de las levaduras enológicas. Fermentan globalmente mal y no poseen los atributos aromáticos. Además, dependiendo de los objetivos y las condiciones de estudio, estas cepas no están siempre adaptadas a los análisis en la fermentación del vino. Por lo tanto, los microbiólogos que abordan estas cuestiones están obligados a menudo a realizar un trabajo equivalente de inactivación de genes en cepas enológicas (sobre todo, sus derivados haploides) para acceder a información fiable sobre el papel de los genes en el proceso de fermentación.
Enfoques de este tipo se han realizado en muchos genes. Muy a menudo los trabajos combinan diferentes enfoques y métodos asociados que permiten obtener una información sobre la expresión genética y la síntesis de la proteína correspondiente. Han sido particularmente útiles para aclarar el papel de los diferentes genes en el caso de familias multigénicas, donde varios genes similares son capaces a priori de realizar las mismas funciones. Estas situaciones son bastante frecuentes y se producen, por ejemplo, para los genes relacionados con la formación de ácido acético y que codifican para las aldehído deshidrogenasas (ALD) o para los genes de transporte de azúcares (HXT). En el caso de los genes ALD, se ha podido mostrar que, entre una familia de 6 genes, uno de ellos, ALD6, tenía un papel clave en la fermentación (Saint Prix et al., 2004). Este tipo de información es valiosa porque puede orientar los análisis y permite saber que las variaciones en la formación de acidez volátil están estrechamente relacionadas con el comportamiento del gen y las propiedades del enzima en cuestión. El transporte de azúcares, glucosa y fructosa, es también una situación ejemplar. La levadura contiene en su genoma más de 20 genes que codifican para los transportadores de hexosas HXT (hexose transporter). Esto es específico de S. cerevisiae y subraya la importancia de esta función para la levadura, que ha desarrollado un sistema complejo para optimizar la fermentación de azúcares presentes en las bayas. La cuestión que se planteó era conocer cuáles eran los transportadores que tenían un papel durante la fermentación alcohólica. El análisis del efecto de la inactivación de toda una serie de genes HXT permitió poner en evidencia que cinco de ellos intervenían en el ciclo fermentativo en fases diferentes, algunos al inicio, y otros para finalizar la utilización de azúcares, y que tenían un impacto variable. Un transportador clave, el más activo en la fermentación del vino, ha sido identificado de esta manera, como HXT3.
Los conocimientos adquiridos sobre los transportadores han resultado posteriormente ser muy útiles para entender las diferencias de comportamiento de las cepas comerciales con respecto a la utilización de azúcares. Se sabe que S. cerevisiae prefiere la glucosa a la fructosa, que por lo tanto es fermentada más tarde. Sin embargo, se habían observado diferencias entre cepas y hacía falta explicar por qué una cepa (Fermichamp) tenía una mejor capacidad de fermentar la fructosa. Dado que el transporte transmembrana es una etapa clave en el metabolismo de azúcares, se avanzó la hipótesis de una modificación del transporte en esta cepa. El análisis detallado de los genes de transporte permitió demostrar a continuación que esta cepa tenía un transportador HXT3 modificado más eficaz para la fructosa (Guillaume et al., 2007). Es evidente que la rápida resolución de esta cuestión no habría sido posible sin el trabajo previo de análisis funcional de los transportadores.
El análisis de la expresión de los genes
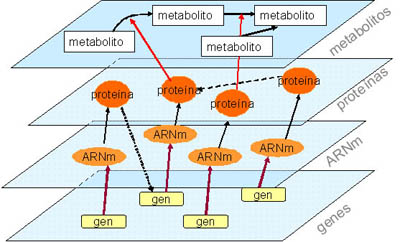
Un aspecto importante de la fisiología de los organismos está relacionado con el control de la expresión génica. Los genes no están todos activos, ni al mismo tiempo ni con la misma intensidad, y su actividad se ajusta en función de las necesidades de la célula. Podemos considerar la expresión de los genes a dos niveles: a nivel de RNA mensajero (o transcrito), o a nivel de proteínas. Las proteínas, la mayoría de las cuales son enzimas, son las entidades activas de la célula que determinan el estado del metabolismo mediante su acción (fig. 1). Sin embargo, se suele evaluar sobre todo la expresión de los genes midiendo los mRNA, porque las herramientas disponibles permiten medir su abundancia a nivel global (el transcriptoma, o sea, el conjunto de transcritos), que no es el caso de las proteínas (para el conjunto de proteínas se habla de proteoma). Desde hace 10 años se dispone de herramientas como los microarrays de DNA (chips de DNA o biochips), que permiten medir simultáneamente la expresión de los 6000 genes de la levadura, en base a la cantidad de mRNA de cada gen. La posibilidad de ver la totalidad del genoma de una levadura «en acción» durante una fermentación alcohólica ha sido un gran avance en la comprensión del comportamiento de la fermentación de la levadura.
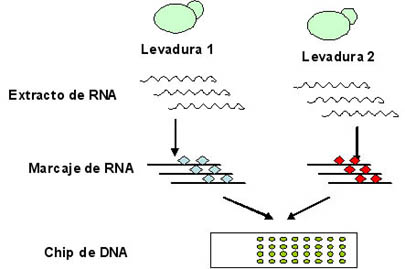
Los chips de DNA se basan todos en el mismo principio. En todos los casos, fragmentos de DNA correspondientes a cada gen son fijados en un soporte de vidrio, con densidades elevadas, de tal manera que se disponen los 6000 genes de la levadura en 3 cm2. La medida de la expresión de los genes se realiza por hibridación de una sonda sintetizada a partir de mRNA extraídos de las levaduras, y que se vuelve fluorescente (fig. 2).
El análisis del transcriptoma primero fue utilizado para explorar la evolución del funcionamiento de los genes durante una fermentación alcohólica (Rossignol et al., 2003). Esto ha permitido ver cuáles son las prioridades de la levadura en fermentación. Por ejemplo, en base a la intensidad de la expresión génica, queda claro que transformar los azúcares en alcohol es una actividad clave de la levadura, pues el conjunto de genes de la vía glucolítica y fermentativa están fuertemente expresados durante todo el proceso. Por ejemplo, estos trabajos han permitido conocer que una de las prioridades de la levadura, una vez inoculada en el mosto, es su adaptación a la desaparición del oxígeno. Esto se traduce en particular en la inducción de genes que le permiten utilizar los esteroles presentes en el mosto, moléculas que la levadura ya no puede sintetizar en anaerobiosis. También ha sido posible precisar cómo la levadura se adapta durante la fermentación a un ambiente cada vez más estresante a causa del agotamiento de nutrientes y de la acumulación del etanol. Las levaduras reaccionan a estos cambios expresando los genes de estrés, cuyas proteínas les confieren una protección contra estas situaciones agresivas. Varios equipos de investigación han evaluado el efecto del nivel de nitrógeno de los mostos sobre la expresión génica, y han demostrado fuertes cambios de algunas vías metabólicas en función del nivel de nitrógeno (Mendes-Ferreira et al., 2007). Otros trabajos han permitido aclarar la respuesta de las levaduras a las bajas temperaturas de fermentación (Zuzuarregui et al., 2006).
La posibilidad de tener una visión global del funcionamiento del genoma ha sido un gran aporte para la comprensión del metabolismo de la levadura que está fermentando. No es menos cierto que las medidas de expresión se limitan sobre todo a la cuantificación de los mRNA. Esto supone un límite importante de estos estudios, pues se sabe que los niveles de proteínas no están siempre perfectamente correlacionados con el RNA correspondiente. Los enfoques «semiglobales» de la medida de las proteínas son posibles mediante la técnica de electroforesis bidimensional. Mediante estos planteamientos, se pueden cuantificar normalmente unas 1000 proteínas. Así pues, sólo es accesible una fracción del proteoma. Muy recientemente han surgido otras técnicas más potentes, pero que por el momento son difícilmente accesibles.
El reto más importante sigue siendo tratar de recopilar la información lo más global posible a los diferentes niveles de funcionamiento de la célula: transcriptoma, proteoma, pero también el metaboloma, o conjunto de metabolitos de la célula. Se trata de ser capaz de integrar los diferentes niveles de análisis para poder representar el funcionamiento del sistema con la ayuda de modelos matemáticos. Entonces se habla de biología de sistemas, que es una disciplina que ha surgido los últimos años y que tiene un futuro prometedor.
La genética para entender las bases de la biodiversidad
Tal como se ha mencionado antes, la comprensión de las bases genéticas de las diferencias en las propiedades de las cepas es un objetivo importante para la mejora de las levaduras. La determinación del origen genético de las propiedades de interés (velocidad de fermentación, producción de compuestos aromáticos, etc.) ha sido muy difícil durante mucho tiempo, sobre todo porque se trata de propiedades complejas, o sea que dependen de la actividad de varios, o incluso de muchos, genes. Aunque las técnicas genéticas que permiten estudiar estas propiedades son utilizadas desde hace tiempo en animales o en plantas (por ejemplo para identificar genes determinantes de la talla de la espiga de trigo o del peso del animal), estas técnicas para levadura se han desarrollado sólo recientemente. Este enfoque, denominado genética cuantitativa, se ha visto también facilitado por la llegada de nuevas herramientas de análisis, como los chips de DNA.
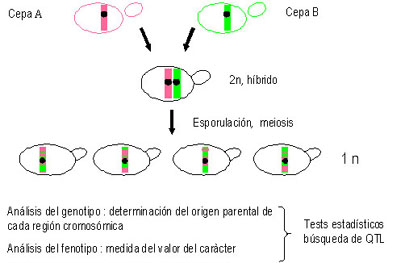
Las estrategias puestas a punto se basan en la identificación de regiones del genoma o QTL (Quantitative Trait Locus, locus de características cuantitativas) que afectan a la característica analizada. Para ello se crea una población recombinante mediante el cruzamiento de dos cepas que tienen valores muy diferentes para un carácter determinado (una de valor alto con otra de valor bajo). Después de la esporulación, se obtiene la población de clones que contienen los genomas de ambos padres totalmente reorganizados (de hecho recombinantes). Entonces, usando herramientas moleculares (marcadores), se procede a identificar el origen parental de cada región del genoma de los clones (fig. 3). Los valores de la característica se miden en todos los individuos y se evalúa la existencia de una relación estadística entre el origen parental de una región del genoma y el valor de la característica. Aunque bastante sencillos en principio, estos procedimientos son realmente complejos y pueden ser difíciles de aplicar según la situación genética analizada. Se han utilizado con éxito para identificar las bases genéticas de diferentes características enológicas (Marullo et al., 2007). Nuestro equipo se ha centrado en las diferencias de rendimiento en fermentación entre una cepa enológica y otra de laboratorio. De esta manera se ha identificado un gen que explica la diferencia de velocidad máxima de fermentación entre la cepa comercial EC1118 y la cepa de laboratorio S288C. Así, hemos podido demostrar que la cepa enológica tiene un alelo más eficaz para un gen de síntesis del p-aminobenzoico, que influye en la velocidad de fermentación mediante el control de la utilización del nitrógeno.
Conclusión
Evidentemente el acceso a los conocimientos sobre el genoma y su funcionamiento ha entrado en una nueva era con la llegada de un arsenal de herramientas y procedimientos cada vez más potentes. El aspecto más notable es la puesta a punto de técnicas de « alto rendimiento », que abarcan desde el análisis de los genomas hasta el estudio de su funcionamiento. El día de mañana, conocer la secuencia de una levadura de vino será una información básica a la que cualquier investigador va a tener fácil acceso. La combinación de estas herramientas y procedimientos actualmente permite a los expertos en genómica y en genética abordar eficazmente viejas preguntas que no tenían respuesta, sobre el comportamiento de la levadura del vino en fermentación. De esta manera se debería ir hacia una mejor comprensión de las bases moleculares de la biodiversidad de las levaduras. Este conocimiento en detalle debe beneficiar en gran medida los procedimientos de selección y de mejora genética de las cepas. En particular, será posible implementar estrategias de hibridación de las cepas con el fin de introducir en las levaduras enológicas las características específicas para una mejor adaptación a las condiciones de vinificación y a la demanda de los enólogos.
Bibliografía
. Goffeau A, Barrel BG, Bussey H, Davis RW, Dujon B, Feldman H, et al. Life with 6000 genes. Science 1996; 274: 546-67.
. Guillaume C. Sablayrolles, J.M. and B. Blondin. Molecular basis of fructose utilization by the wine yeast Saccharomyces cerevisiae: a mutated HXT3 allele enhances fructose fermentation. Applied and Environmental Microbiology 2007; 73: 2432-9.
. Liti G, et al. Population genomics of domestic and wild yeasts. Nature 2009; 458: 337-41. Marks VD, van der Merwe GK, van Vuuren HJ. Transcriptional profiling of wine yeast in fermenting grape juice: regulatory effect of diammonium phosphate. FEMS Yeast Research 2003; 3: 269-87.
. Marullo P, Aigle M, Bely M, Masneuf-Pomarède I, Durrens P, Dubourdieu D, Yvert G. Single QTL mapping and nucleotide-level resolution of a physiologic trait in wine Saccharomyces cerevisiae strains. FEMS Yeast Research 2007; 7: 941-52.
. Mendes-Ferreira A, Del Olmo M, García-Martínez J, Jiménez-Martí E, Mendes-Faia A, Pérez-Ortín JE, Leão C. Transcriptional Response of Saccharomyces cerevisiae to Different Nitrogen Concentrations during Alcoholic Fermentation. Applied and Environmental Microbiology 2007; 73: 3049-60.
. Novo et al. Eukaryote-to-eukaryote gene transfer events revealed by the genome sequence of the wine yeast Saccharomyces cerevisiae EC1118. Proceedings National Academy Sciences USA 2009; 106: 16333-8.
. Rossignol T, Julien A, Dulau L, Blondin B. Genome-wide monitoring of wine yeast gene expression during alcoholic fermentation. Yeast 2003; 20: 1369-85.
. Saint-Prix F, Bönquist L, Dequin S. Functional analysis of the ALD gene family of Saccharomyces cerevisiae during anaerobic growth on glucose: the NADP+-dependent Ald6p and Ald5p isoforms play a major role in acetate formation. Microbiology 2004; 150: 2209-20.
. Zuzuarregui A, Monteoliva L, Gil C, Del Olmo M. Transcriptomic and proteomic approach for understanding the molecular basis of adaptation of Saccharomyces cerevisiae to wine fermentation. Applied and Environmental Microbiology 2006; 72: 836-47.