Introducción
Una empresa local productora de vinos facilitó que nuestros docentes y alumnos de la Cátedra de Industria y Tecnología V (Fermentaciones) de la Carrera de Alimentos pudieran realizar experiencias a escala piloto usando parámetros industriales. Se decidió entonces obtener vinos propios utilizando cepas disponibles en el mercado de Saccharomyces cerevisiae var. bayanus, var. ellipsoides y flora nativa.
El objetivo de este estudio parte de un trabajo de investigación en una matriz alimenticia alcohólica1 en el que se determinaron los aminoácidos libres y amonio más representativos de muestras nacionales e internacionales del mercado local. (Acerca de la determinación de aminoácidos en fermentaciones alcohólicas, hacemos mención de un excelente artículo.2)
Nuestra intención desde el inicio de la experiencia fue verificar el comportamiento allí descrito en una nueva matriz alcohólica como son los vinos.
En el diseño experimental se consideraron 5 semanas para obtener dichos vinos partiendo de uvas merlot, provenientes de la provincia de San Juan, y torrontés de la provincia de Salta. Las uvas adquiridas tenían un alto grado de maduración.
Se tomaron muestras representativas a tiempo 0 y durante las siguientes 4 semanas (una por semana). Los parámetros esenciales de la fermentación fueron controlados con metodología oficial del Instituto Nacional de Vitivinicultura de la Argentina. Estos parámetros fueron: pH, acidez total, azúcares reductores, sulfito libre y grado alcohólico.
Nuestro interés por los aminoácidos en este estudio enológico se debe a:
- por ser la fuente de nitrógeno más importante de asimilación de la levadura; es decir que los aminoácidos son nutrientes fundamentales para el desarrollo y reproducción del organismo durante la fermentación alcohólica;
- porque la velocidad de fermentación dependerá de la calidad y cantidad de aminoácidos en el mosto. Además, son precursores de las propiedades organolépticas del vino al transformarse en alcoholes, aldehídos, ésteres, ácidos dicetónicos, etc. (principales responsables del flavor);3
- como criterio de genuinidad, es decir de no adulteración, los aminoácidos están desplazando (en diferentes países) a otros criterios hasta ahora usados, como son los pigmentos (por ejemplo antocianinas), presentes originalmente en la uva y en el vino, por ser más estables, seguros y confiables;
- si hay baja calidad y cantidad de aminoácidos habrá baja población de levaduras y por lo tanto posibilidad competitiva de otros microorganismos. Por el contrario, un exceso en cantidad producirá un gran crecimiento de levaduras afectando el aroma del vino;
- el contenido en aminoácidos de las uvas es dependiente de las condiciones climáticas, el cultivo, los tratamientos del suelo y las prácticas realizadas sobre el mismo.3
En fin, concluimos que conocer estos parámetros analíticos cualitativa y cuantitativamente podrían ser tomados en cuenta como índices de genuinidad o huella dactilar de los vinos producidos.
Con respecto a la metodología analítica que utilizamos, la clásica es la determinación por RP-HPLC de derivatización. Consultada la bibliografía internacional sobre el tema, nos encontramos solamente con una referencia (Carnevillier et al.4) que presenta una síntesis de los diversos derivatizantes usados: OPA, FMOC, cloruro de dansilo, PITC, etc. Existe una referencia similar a la metodología usada por nosotros.6
También se determinó la proteína existente en cada muestra del experimento. Así, los principales objetivos del presente trabajo son dos:
- la identificación del perfil de aminoácidos libres de 3 varietales argentinos, ya que estos analitos desempeñan un importante papel en el progreso de la fermentación y en la formación bioquímica de alcoholes de alto peso molecular, y
- tratar de caracterizar los vinos argentinos por el conocimiento de sus perfiles aminoacídicos (aminograma).
A continuación se muestran los resultados obtenidos, con la advertencia que se circunscriben a las muestras originales tomadas en Argentina, destacando al respecto que varios países están trabajando intensamente con el fin de tipificar sus producciones y asegurar la mejor calidad (Francia, Chile, Alemania, Italia, etc.).3
Material y método
Uvas
Las materias primas fueron compradas (inicios de abril) en el mercado local. Como fue indicado previamente, las uvas estaban muy fermentadas. Se realizó un recuento de población inicial y se encontró 40 millones de UFC/mL para merlot y de 10 millones de UFC/mL para torrontés.
Se colocaron en cada tanque de fermentación de la empresa local, aproximadamente, 100-110 kg de uva de los que se obtuvieron 60-70 L de mosto y se siguió el protocolo que a continuación se detalla:

Toma de la muestra y preparación para el análisis
A partir de la primera semana, el hollejo se separa (sin moverlo) para permitir entrar el sacamuestras hasta el fondo del tanque. Se obtuvieron muestras por duplicado para poder realizar todos los análisis previamente descritos. Las muestras tomadas para determinación de los aminoácidos fueron congeladas a – 20 ºC hasta su análisis.
Las mismas fueron filtradas por papel de filtro SS banda azul para retener el material grueso. A cada filtrado se le agregaron 2 gotas de hidróxido de sodio (1 N/mL de filtrado para obtener un pH aproximado a 6). Cuando no fue neutralizado y el pH del mosto era bajo no se pudo lograr la acción del búfer del reactivo de derivatización.
Las muestras se filtraron por filtros Millex LCR 13 y se tomaron 200 µL de muestra a las que se les agregó 4 µL de estándar interno AABA 2,5 mM más 800 µL de agua.
Se tomaron 10 µL de dilución anterior a la que se le agregó 70 µL de búfer y 20 µL de reactivo Accutag. Se voltea, se calienta a 50 ºC durante 5 min y se inyectan 5 µL de aminoácidos derivatizados.
Las referencias internacionales4-6 recomendaban que la muestra fuera desproteinizada con etanol, luego centrifugada y finalmente rotoevaporada para eliminar el solvente, y finalmente filtrar por membrana con cut-off 1000. Pero esta técnica fue descartada por ser más cara que la que finalmente usamos.
Preparación del estándar de aminoácidos
Se partió del kit Accutag del que se tomaron 200 µL del estándar de aminoácidos 2,5 mM (cistina está 1,25 mM) a la que se le agregó 4 µL de Estandar Interno AABA (ácido alfa-aminobutírico) 2,5 mM más 800 µL de agua. Se tomó 10 µL de la dilución y se procedió como en la muestra.
Inyectar 5 µL representa 50 pmol de cada aminoácido, menos la cistina que está a 25 pmol. El orden de elución es el siguiente: AMQ, ácido aspártico, serina, ácido glutámico, glicina, histidina, amoníaco, arginina, treonina, alanina, prolina, cistina, tirosina, valina, metionina, lisina, isoleucina, leucina y fenilalanina.
- Reactivos:
- Acetonitrilo grado HPLC (J.T. Baker).
- Agua calidad HPLC: obtenida por filtración con Milli-Q.
- Todos los solventes fueron filtrados diariamente por filtros Millipore GVWP 04700.
- Estándar interno AABA (Sigma).
- El kit AccQ-Tag es marca registrada de Waters Corporation (D’Amico Sistemas, Buenos Aires).
- Instrumental:
- Cromatógrafo líquido Agilent Modelo 1050 compuesto por: bomba cuaternaria, desgasificador en línea por vacío, inyector automático y horno para la columna, conectores para microbore.
- Detector fluorimétrico Modelo 1046 con celda de 1 µL.
- Integrador HP 3396 II.
- Condiciones cromatográficas:
- Columna Hypersil ODS de 200 mm x 2,1 mm. El diámetro de partícula es de 5 µm y el diámetro de poro es de 12 ηm. Se usó guardacolumna de 3,9 x 20 mm rellena con fase reversa, compatible con la columna analítica.
- Gradiente ternario compuesto por la línea A de búfer con pH 5,05; la línea B es acetonitrilo y la C es agua HPLC.
- El gradiente ternario fija la corrida en aproximadamente 30 min, incluyendo el reequilibrio de la columna.
- El uso de reactivos de baja calidad es una fuente de contaminación del gradiente y, por lo tanto, de aparición de picos espurios.
- Si se trabaja a pH 4,95 se mejora notablemente la resolución del AMQ/Asp (esto es importante en la detección UV, no así en la fluorimétrica usada por nosotros).
- Si el pH del búfer está en 5,80 se mejora la resolución de serina y asparragina.
- Disminuyendo la pendiente del gradiente entre 0,5 y 18 min, se mejora la resolución general pero se desmejora la resolución Gli/Hist y Arg/Treo.
- Flujo: 0,33 mL/min.
- Longitud de onda de excitación: 250 ηm.
- Longitud de onda de emisión de la fluorescencia: 395 ηm.
- Temperatura de la columna: 40 ºC.
- Volumen de inyección: 5 ηL.
La identificación se hizo sobre la base de los tiempos de retención y al orden de elución indicado previamente.
Validación del método
La repetibilidad fue examinada mediante 5 inyecciones consecutivas de la misma muestra en el día de trabajo. La tabla 1 muestra los desvíos estándar obtenidos para los aminoácidos principales.
La reproducibilidad en diferentes días también fue examinada inyectando los mismos estándares cinco veces en el período de 2 semanas. En todos los casos, el coeficiente de variación de las concentraciones calculadas de los aminoácidos más importantes fue menor al 5%.
La linealidad de la respuesta fluorimétrica se constató usando 4 concentraciones diferentes de los aminoácidos estándares, de modo que cubrieran los niveles de los aminoácidos más importantes en uvas, mostos y vinos. Se construyeron curvas de calibración para los aminoácidos más importantes.
El análisis de regresión lineal mostró coeficientes de correlación (r) entre 0,95 y 0,98. El cálculo de las concentraciones de aminoácidos se realizó en base al estándar interno AABA y a su constancia en los resultados obtenidos. Se controló también con relación a las concentraciones de los estándares externos (2,5 mM y 1,25 mM, respectivamente).
Resultados
Datos de azúcares reductores, sulfito y acidez
Al finalizar la primera semana de iniciada la fermentación se obtuvieron los siguientes datos:
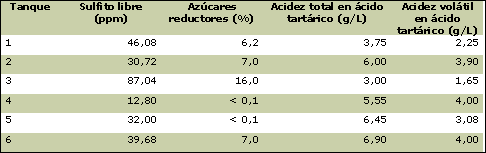
A la segunda semana de iniciada la fermentación se agregan 40 g de sulfito de sodio a los tanques 4 y 5 (para poder conservarlo ya que finalizó la fermentación y pretendemos bloquear la fermentación secundaria o maloláctica y también evitar el crecimiento de posibles hongos). Los resultados obtenidos son:
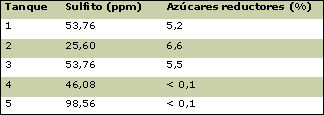
A la tercera semana de iniciada la fermentación:
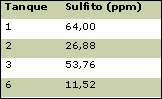
Controles realizados del sulfito libre
A la quinta semana de iniciado el proceso, los resultados fueron:
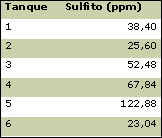
A la séptima semana de iniciada la fermentación:
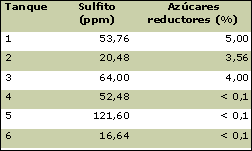
A la undécima semana de iniciada la fermentación:
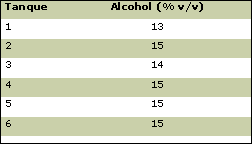
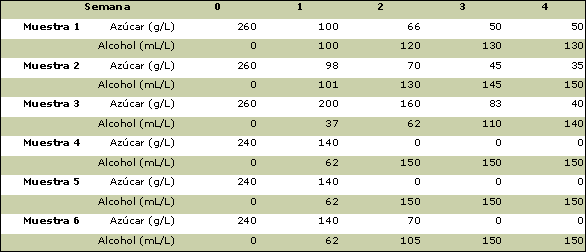
Datos obtenidos de la variación de alcohol y de azúcar durante la vinificación
Datos obtenidos de aminoácidos
La tabla 1 presenta los resultados comparativos de la investigación, expresados como rango en ppm, de todos los aminoácidos (n = 5) presentes en los vinos blancos (torrontés) para los diferentes tiempos de elaboración previamente definidos.
(Hacemos notar que se ha estudiado solamente la precisión de los aminoácidos más significativos.)
La tabla 2 presenta los resultados comparativos de la investigación, expresados como rango en ppm, de todos los aminoácidos (n = 5) presentes en el vino tinto (merlot) para los diferentes tiempos de elaboración. (Como ya se ha indicado, se ha estudiado sólo la precisión de los aminoácidos más significativos.)
Los valores de aminoácidos encontrados (tablas 1 y 2) son:
TORRONTÉS
• Uva: 2850 ppm (58,6% es prolina)
• Mosto:
S. ellipsoides: 2410 ppm (68,6% Pro) → 5869 ppm (91,3% Pro) → 5716 ppm (92,3% Pro)
S. bayanus: 4957 ppm (90,8% Pro) → 4789 ppm (92,2% Pro) → 5716 ppm (92,3% Pro)
Nativa: 6980 ppm (92,9% Pro) → 5741 ppm (92,0% Pro) → 5582 ppm (92,9% Pro)
• Vino:
S. ellipsoides: 4944 ppm (88,9% Pro)
S. bayanus: 4726 ppm (90,6% Pro)
Nativa: 5713 ppm (92,0% Pro)
MERLOT
• Uva: 8452 ppm (91,9% es prolina)
• Mosto:
S. ellipsoides: 5940 ppm (87,5% Pro) → 4460 ppm (89,9% Pro) → 4399 ppm (89,9% Pro)
S. bayanus: 5962 ppm (86,9% Pro) → 4709 ppm (91,3% Pro) → 4166 ppm (88,4% Pro)
Nativa: 5800 ppm (86,4% Pro) → 4090 ppm (89,2% Pro) → 3749 ppm (91,4% Pro)
• Vino:
S. ellipsoides: 4876 ppm (90,3% Pro)
S. bayanus: 4853 ppm (92,4% Pro)
Nativa: 4874 ppm (92,5% Pro)
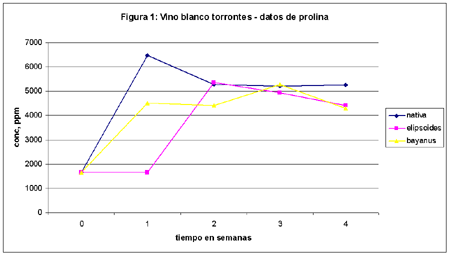
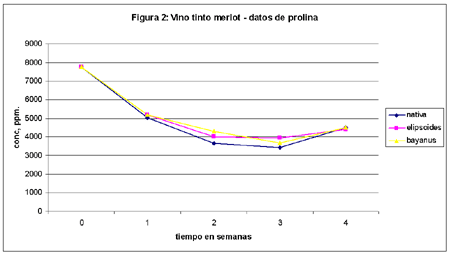
En las figuras 1 y 2 se muestran los datos de concentración (expresados en ppm), de prolina (torrontés y merlot, respectivamente) en función del tiempo para las tres levaduras usadas.
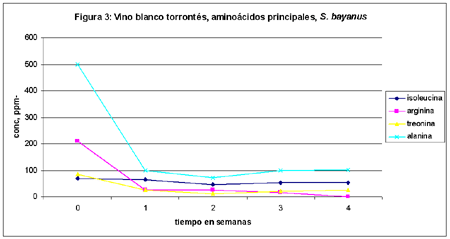
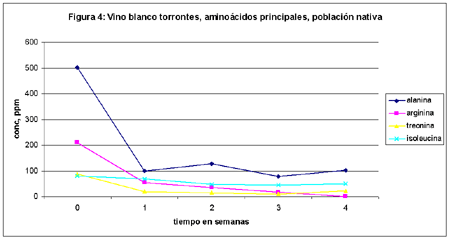
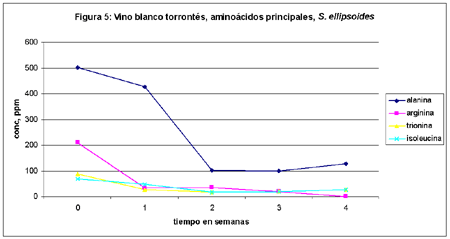
En las figuras 3, 4 y 5 se muestran la variación de concentración (en ppm) de los aminoácidos más importantes encontrados en torrontés para Saccharomyces var. bayanus, var. ellipsoides y flora nativa, respectivamente.
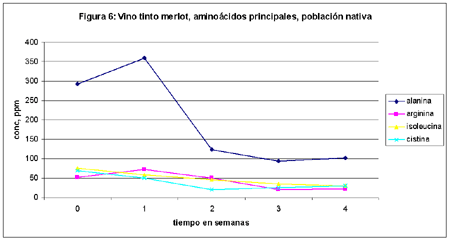
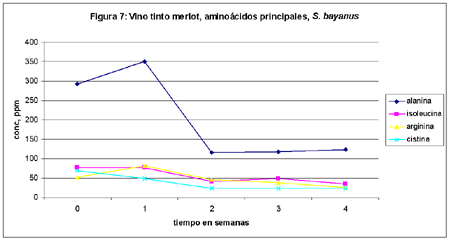
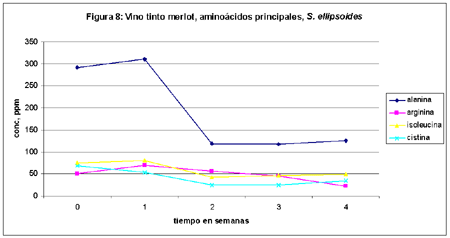
En las figuras 6, 7 y 8 se muestran los mismos parámetros para merlot.
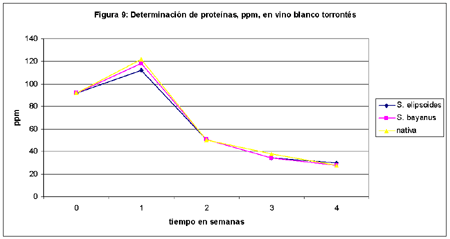
La figura 9 presenta los datos de la concentración (en ppm), de proteínas en vinos blancos y su evolución durante el proceso (n = 5 y CV = 4,2%).
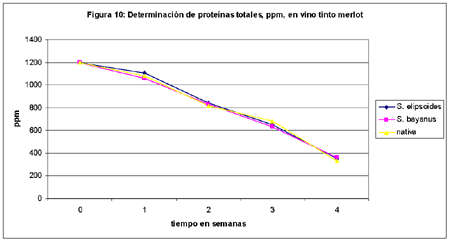
En la figura 10 se observan los datos de la concentración (en ppm), de proteínas en vinos tintos y su evolución durante el proceso (n = 5 y CV = 4,9%).
La técnica oficial para la determinación de las proteínas en vinos es el Kjeldahl. Se usó el método del rojo de pirogalol (PROTIU/LCR) de Wiener Laboratorios (Rosario, República Argentina), previo contraste con Kjeldahl, debido a que es una metodología que determina proteínas en muy bajas concentraciones. Otras metodologías probadas fueron Folin-Ciocalteau y Meulemanns. Para el primero de ellos, al precipitar la proteína con el ácido tricloroacético, éste arrastraba junto con la misma otros compuestos adsorbidos. Fue, por tanto, desechado, ya que al reconstituirse el precipitado se obtenían resultados poco reproducibles. Meulemanns, por el contrario, fue descartado especialmente en los vinos tintos, ya que las antocianinas cambiaban fuertemente de color al agregarle el reactivo.
Estudio de las interferencias debidas al efecto matriz
Se realizaron adiciones estándar a diferentes matrices de vinos blancos. Para ello se prepararon soluciones estándar de los principales aminoácidos y se añadieron (0-20-40-60 µL) a series de 250 µL de muestras de vino.
Todas las muestras fueron derivatizadas con Accutag por triplicado y después separados los aminoácidos como fue descrito previamente. Los resultados fueron:
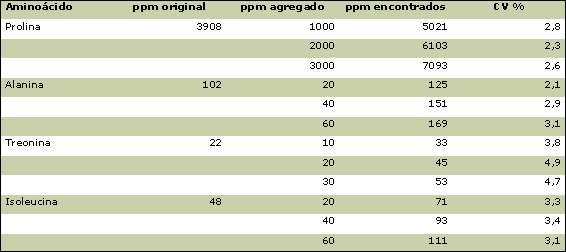
Como se observa, el CV es menor del 5%. De esta forma podemos asegurar que la exactitud es buena y que no hay interferencias debidas a la matriz.
Conclusiones
1. Prolina es el aminoácido (en realidad iminoácido) predominante en el producto final. Se cumple perfectamente lo predicho por Margaret Jones en la matriz cerveza,2 es decir que no es metabolizado por ninguna especie de levadura (grupo D) en condiciones de anaerobiosis. De todos modos hay un consumo inicial y luego se produce la meseta.
2. Surge a continuación la pregunta de por qué en el vino blanco los valores de este aminoácido ascienden para después buscar la meseta como en el merlot. Es más lógico lo que ocurre con esta última en donde de un valor inicial alto el proceso fermentativo lleva a la prolina a buscar una meseta. Deducimos que lo que ocurre en el torrontés es una consecuencia del sulfito agregado y a la lisis parcial de la población nativa. En el futuro seguiremos trabajando este tema.
3. El segundo aminoácido en importancia cuantitativa es alanina: esto también coincide con la bibliografía existente,2 ya que este aminoácido pertenece al grupo C, es decir a los analitos que son absorbidos por la levadura después de un período de latencia.
4. Los aminoácidos que aparecen en tercer nivel son isoleucina y arginina (en ambos vinos), treonina (blanco) y cistina (tinto): todos ellos pertenecen al grupo B:2 la levadura los absorbe lentamente.
5. La fuente de aminoácidos en el mosto son los polipéptidos producto de la proteólisis de las proteínas presentes inicialmente en la uva. Vitis vinifera contiene aproximadamente 500-1500 ppm de proteína. La proteína residual en el vino es del orden de 50-200 ppm.7 En el vino blanco, los valores de proteínas están dentro de lo que dice la referencia, mientras que en el tinto es aproximadamente 10 veces mayor, y ello es debido a que los suelos fueron extra fertilizados y a la edad del viñedo (datos aportados por el productor de la uva). En el futuro profundizaremos este aspecto.
6. La bibliografía internacional3-6 estipula que, en los vinos, los aminoácidos libres, determinados por técnicas cromatográficas varias, se encuentran en las siguientes concentraciones: prolina: 700-1500 ppm; alanina: 70-150 ppm; arginina: 20-150 ppm; isoleucina: 10-80 ppm; cistina: 10-100 ppm; treonina: 20-60 ppm. Nuestros resultados son bastante concordantes con las referencias internacionales salvo el caso de prolina en donde hallamos 10 veces más en los mostos tintos (explicable por el mayor porcentaje de proteína encontrada) y cerca de ocho veces más en los mostos blancos. Creemos que ello se debe a la sensibilidad del método para cuantificar a este aminoácido. En las referencias bibliográficas citadas, ninguno de los métodos usados tiene la sensibilidad tan alta para la prolina (medida en femtomoles de aminoácidos), inclusive se usan metodologías que no lo cuantifican como es la metodología OPA.3
7. Como venimos afirmando desde que comenzamos a trabajar con aminoácidos el tema de genuinidad,1,8-10 si se conocen los parámetros tecnológicos del proceso y se correlaciona con los aminoácidos libres presentes, será posible fijar la calidad del producto obtenido. Por ello consideramos que los aminoácidos son huellas dactilares importantes e inclusive pueden desplazar a las antocianinas como se viene haciendo en la actualidad. Recordar que no sólo se debe tener en cuenta lo predicho, sino que también se deberá considerar las relaciones clima, suelo, abonado, lugar geográfico, edad de los viñedos, el papel de los aminoácidos como precursores de aminas biógenas, etc.
8. Además de todo lo dicho previamente, la presencia/ausencia de estos analitos fijará también el flavor del vino por las transformaciones que sufren los aminoácidos, dando alcoholes superiores muy particulares, aldehídos, ésteres, ácidos dicetónicos, etc.
9. El contenido en aminoácidos libres del mosto y de las uvas depende fuertemente de la variedad de uva usada y, al mismo tiempo, del tipo de levadura: la nativa consume menos prolina que las otras.
10. En el futuro produciremos vino usando el mismo proceso pero cambiando de zonas geográficas argentinas, estadios madurativos, etc.
Bibliografía
1. Giraudo M., Sanchez Tuero H., Pomilio A.,Pavesi R., Markowski I., Guirin G., Montesano J.: «Determination of free amino acids and ammonium in blonde and black beers and malts of the argentinian market by rp-hplc with fluorescent detection», Alimentaria 2004; 359: 85-89.
2. Pierce: The Margaret Jones memorial lecture: amino acids in malting and brewing, J Int Brew 1982; 88: 228.
3. Soufleros E., Bouloumpasi E., Tsarchopoulos L.: Primary amino acid profiles of Greek white wines and their use in classification according to variety, origin and vintage, Food Chemistry 2003; 80: 261-273.
4. Carnevillier V., Schlich P., Guerreau J.: Characterization of the production regions of Chardonnay wines by analysis of free amino acids, Vitis 1999; 38: 37.
5. Hernández Orte P., Guitart A., Cacho J.: Amino acids determination in musts and wines by hplc after derivatization with phenlisothiocyanate, Am J Enol Vitic 1997; 48: 229.
6. Hernández Orte P., Ibarz M., Cacho J.: Amino acids determination in grape juices and wines, Chromatographia 2003; 58: 29-35.
7. CENEXA, Tablas de composición química, 1995.
8. Giraudo M., Sanchez Tuero H., Muset G., Pavesi R., Castañeda R., Fernández M., Noseda D., Markowski I., Guirin G.: Determinación cuantitativa de aminoácidos en quesos reggianito argentino por derivatización con 6-AQC y rp-hplc, Alimentaria 2002; 337: 121-126.
9. Giraudo M., Sanchez Tuero H., Pavesi R., Markowski I., Guirin G., Montesano J.: El proceso productivo de los jugos cítricos y la determinación de los aminoácidos libres por rp-hplc en jugos frescos y concentrados como criterio de genuinidad, Alimentaria 2004; 350: 97-102.
10. Asensio M., Valdés E.: Characterization of some spanish white grape wine cultivars by morphology and amino acids analysis, Scientia-Horticulturae 2002; 93 (3/4): 289-299.
11. Lehtonen P.: Determination of amines and amino acids in wine. A review, Am J Enol Vitic 1996; 47: 127-133.